Otimizando pipelines que envolvem dados desbalanceados
Utilizaremos o framework tidymodels para machine learning em R com o auxílio do pacote workflowsets para otimizar pipelines de dados desbalanceados

O problema envolvendo dados desbalanceados
A tarefa de classificação com dados desbalanceados é muito comum na vida real podendo variar desde um leve viés até um enorme desequilíbrio na distribuição da classe de interesse. Problemas mais comuns envolvem:
- Detecção de fraude;
- Previsão de inadimplência;
- Identificador de spam;
- Busca por anomalias/outliers;
- Detecção de possíveis roubos/furtos/vulnerabilidades;
- Previsão de churn;
- etc
Este tipo de tarefa representa um enorme desafio para modelagem preditiva pois a maioria dos algoritmos de machine learning foram projetados sob suposição de haver um número igual de exemplos para cada classe de interesse.
E isso é um grande problema pois normalmente estamos interessados em prever a classe minoritária e para isso é preciso tomar uma série de decisões, como por exemplo: métrica utilizada, método para validação cruzada, adoção (ou não) do uso de métodos de reamostragem, quais algoritmos utilizar, qual será o threshold, etc

Lidar com dados desbalanceados é um assunto longo portanto tentarei dar mais atenção apenas em um hack para encontrar a melhor forma de se aplicar o balanceamento dos dados. Não pretendo me aprofundar na teoria envolvida na escolha das métricas neste post, caso o leitor deseje se aprofundar sobre a teoria envolvida com classificação que envolve dados desbalanceados, sugiro a leitura do livro: Imbalanced Classification with Python - Choose Better Metrics, Balance Skewed Classes and Apply Cost-Sensitive Learning e consultar os links de referência no final do post).
Objetivo
Utilizaremos neste post o pacote workflowsets a fim de otimizar o pipeline de reamostragem da base para lidar com o desbalanceamento dos dados.
Para efeitos de comparação, utilizarei como referência o (excelente) post escrito recentemente pela Julia Silge em seu blog que também aborda o problema de dados desbalanceados utilizando um conjunto de dados de uma competição do Kaggle. Utilizarei a mesma configuração de pré-processamento adotado em seu post para que a comparação seja justa.
Portanto, nosso objetivo de modelagem será prever se uma colisão com animais selvagens resultou em danos a aeronave.
⚠️ Este dataset é rico em possibilidades para diferentes tipos de pré processamentos e por isso convido o leitor a analisá-lo com maior profundidade e também a compartilhar seus resultados!
Dependências
Primeiro vamos carregar as bibliotecas necessárias e algumas funções desenvolvidas para o post
library(tidyverse) # ds toolkit
library(tidymodels) # ml toolkit
library(baguette) # bag_tree
library(themis) # imbalanced
library(workflowsets) # opt pipelines
library(patchwork) # arrange plots
doParallel::registerDoParallel()
theme_set(theme_bw())
(Clique aqui para ver as funções print_table e conf_mat_plot importadas)
# Para o print de tabelas
print_table <- function(x, round=0, cv=F, wf=F, bm=F, ...){
if(round>0) x <- x %>% mutate_if(is.numeric, ~round(.x, round))
if(cv==T){
columns_spec = list(
.metric = reactable::colDef(minWidth = 75),
.estimator = reactable::colDef(minWidth = 70),
.config = reactable::colDef(minWidth = 120)
)
} else if(wf==T){
columns_spec = list(
wflow_id = reactable::colDef(minWidth = 100),
.metric = reactable::colDef(minWidth = 100),
preprocessor = reactable::colDef(minWidth = 110),
rank = reactable::colDef(minWidth = 50),
n = reactable::colDef(minWidth = 50)
)
}else if (bm==T){
columns_spec = list(
wflow_id = reactable::colDef(minWidth = 130),
model = reactable::colDef(minWidth = 80)
)
}else{
columns_spec = NULL
}
reactable::reactable(x, striped = T, bordered = T,
highlight = T, pagination = F, resizable = T,
columns = columns_spec, ...)
}
# Para plot da matriz de confusao e distribuicoes de probabilidade
conf_mat_plot <- function(x, null_model = FALSE){
p1 <-
x %>%
select(.pred_class, damaged) %>%
table() %>%
conf_mat() %>%
autoplot(type = "heatmap")+
labs(title = "Matriz de confusão")
p2 <-
x %>%
ggplot() +
geom_density(aes(x = .pred_damage, fill = damaged),
alpha = 0.5)+
labs(title = "Distribuições de probabilidade previstas",
subtitle = "por classe")+
scale_x_continuous(limits = 0:1)+
scale_fill_brewer(palette="Set1")
p1 | p2
}
Em seguida vamos importar os dados provenientes da competição Inclass do Kaggle SLICED s01e02 - Predict whether an aircraft strike with wildlife causes damage. Para mais informações consulte a documentação e dicionário dos dados.
df <- read_csv("train.csv")Note que carregamos apenas os dados de treino pois os dados de teste não possuem a target.
Preparar dados
Tratar a variável target damaged e avaliar sua distribuição:
df <- df %>%
mutate(damaged = if_else(damaged==1, "damage", "not_damage") %>%
factor(levels = c("damage", "not_damage")))(Clique aqui para ver o código do gráfico abaixo)
p1 <- df %>%
count(damaged) %>%
ggplot(aes(x=rev(damaged), y=n, fill=damaged))+
geom_bar(stat = "identity")+
scale_fill_brewer(palette="Set1")+
theme(legend.position = "bottom")+
labs(y="Número de instâncias", x = "")
p2 <- df %>%
count(damaged) %>%
arrange(desc(damaged)) %>%
mutate(prop = n / sum(n)) %>%
mutate(ypos = cumsum(prop)- 0.5*prop )%>%
ggplot(aes(x="", y=prop, fill=damaged)) +
geom_bar(stat="identity", width=1) +
coord_polar("y", start=0) +
theme_void() +
theme(legend.position="none") +
geom_text(aes(y = ypos,
label = paste(scales::comma(n, big.mark = "."),
scales::comma(n/sum(n), big.mark = ".",
suffix = "%" ),sep = "\n")
),
color = "white", size=6) +
scale_fill_brewer(palette="Set1")
p1 + p2 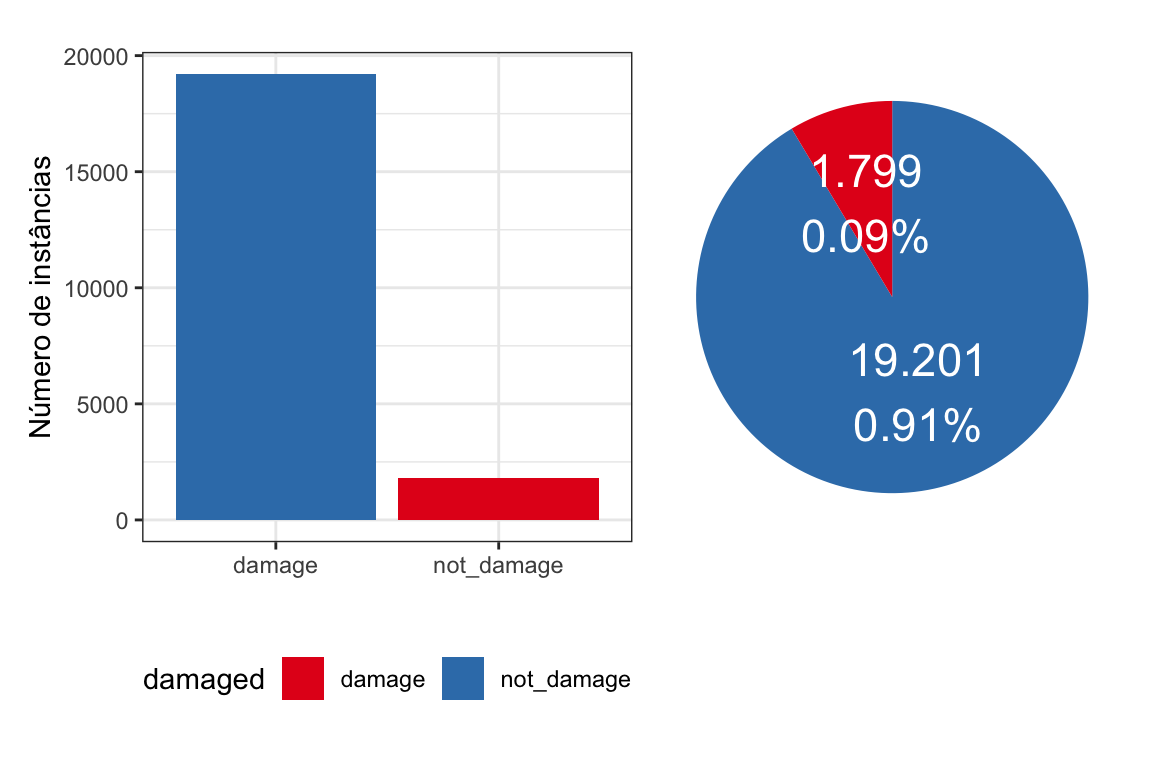
Veja que estamos diante de um problema que existem aproximadamente 9 casos de dano para cada 100 eventos observados.
Breve análise exploratória
Vamos iniciar a exploratória com uma avaliação geral dos dados brutos
DataExplorer::plot_intro(df, ggtheme = theme_bw(),
theme_config = list(legend.position = "bottom"))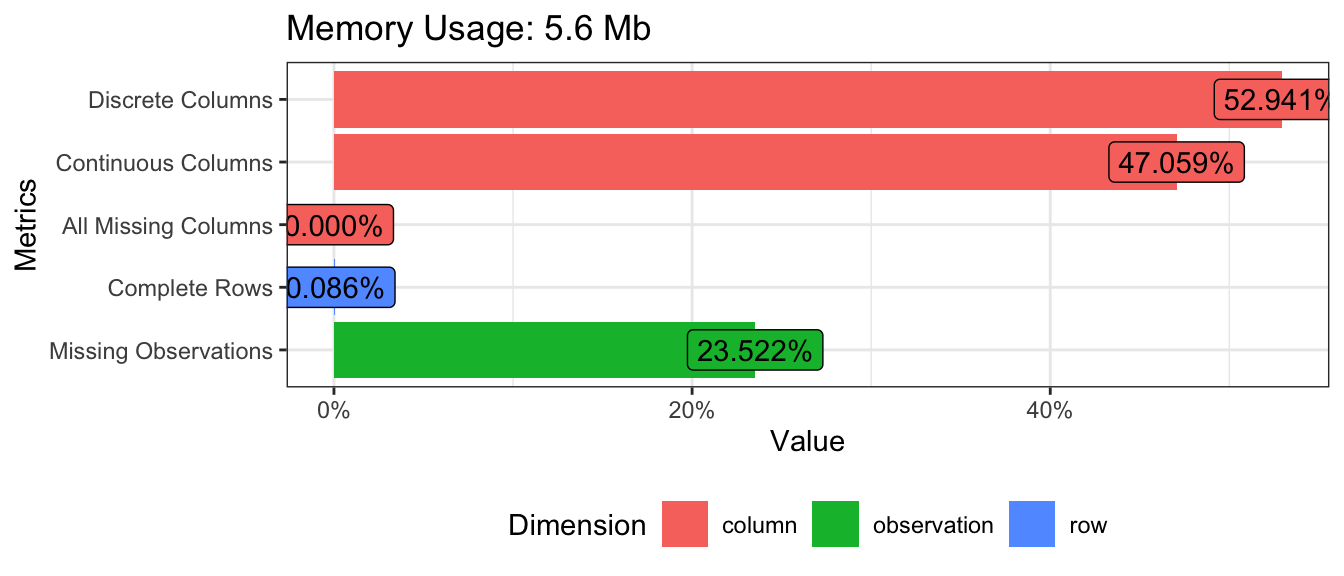
Primeira informação que chama atenção é que quase 1/4 desses dados é faltante. Vamos olhar a estrutura dessa base de maneira mais aprofundada:
df %>%
sample_frac(0.01) %>%
visdat::vis_dat()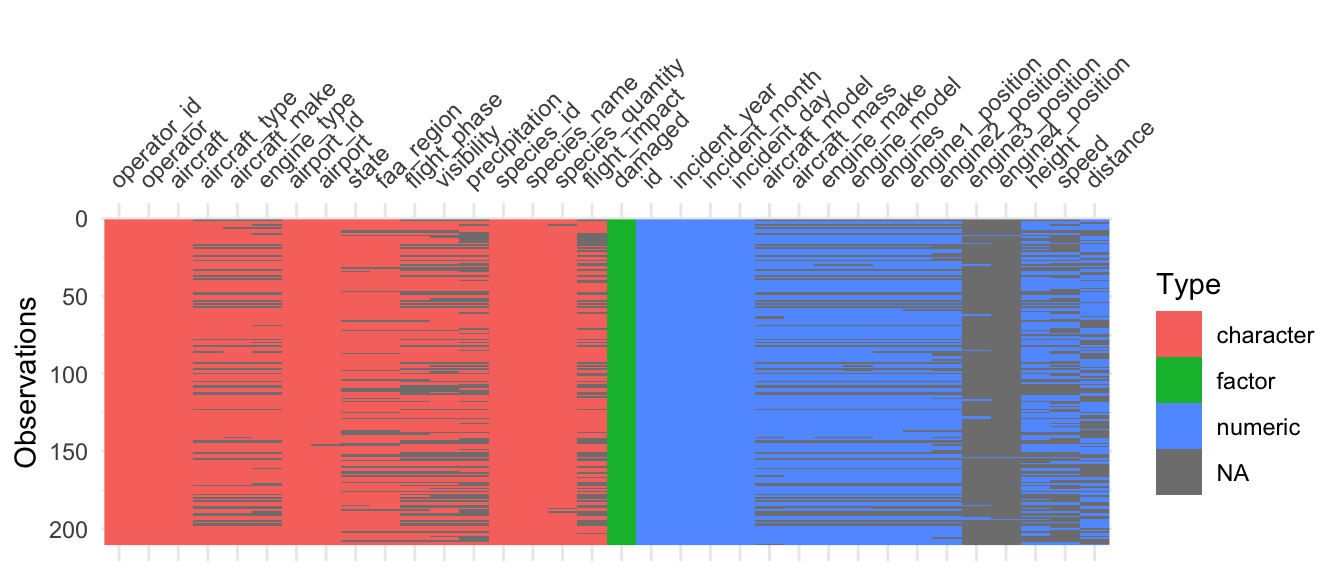
Parece existir algum padrão nos dados faltantes (que coocorrem em diveros atributos). Além disso algumas colunas estão quase inteiramente vazias e serão descartadas no processo de modelagem.
Uma visão geral das classes das features categóricas:
df %>%
select(-damaged, -id)%>%
mutate_all(as.factor) %>%
inspectdf::inspect_cat() %>%
inspectdf::show_plot()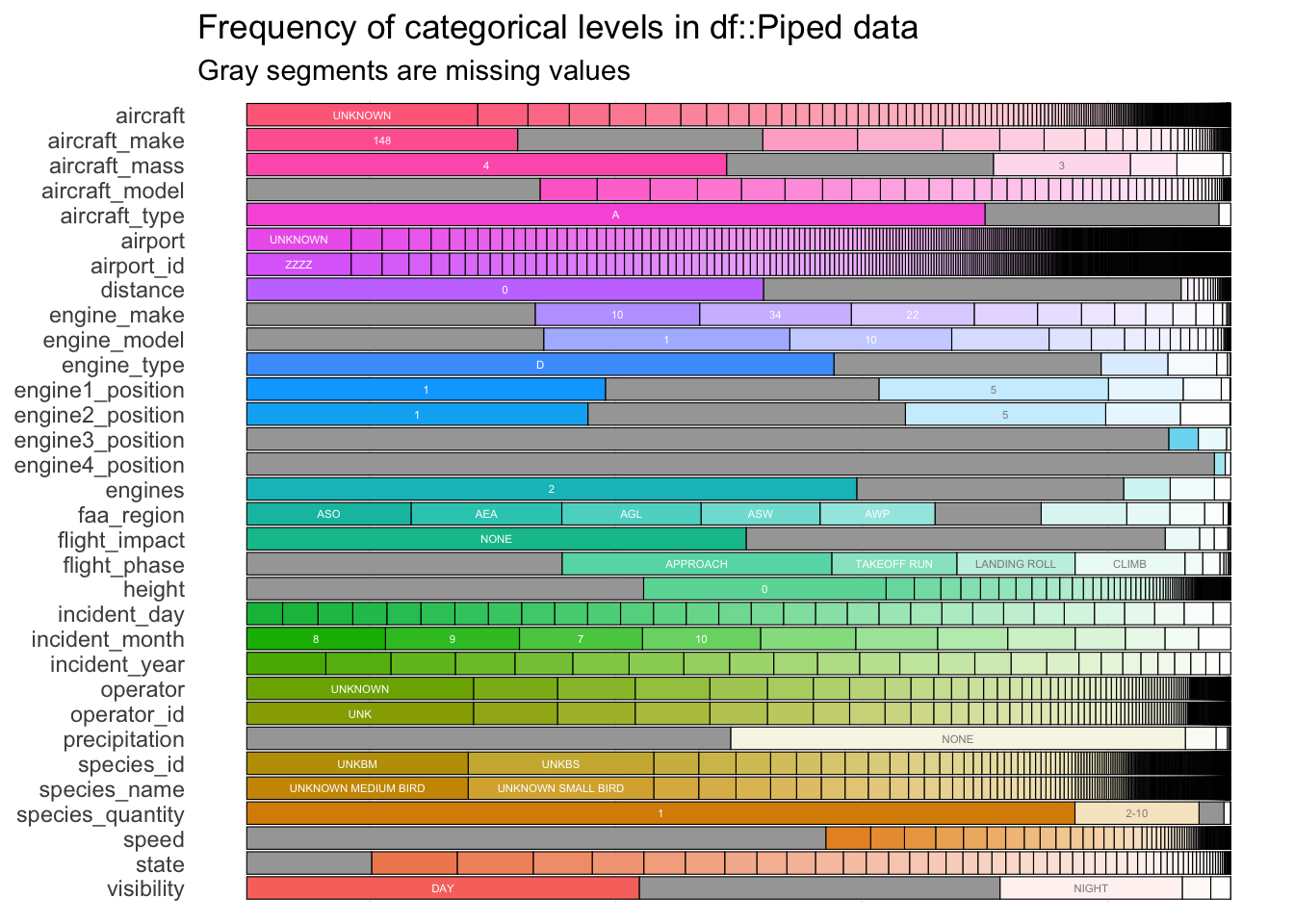
Algumas features possuem muitas classes e caso seja feita a transformação one-hot-encoding (estratégia amplamente utilizada para lidar com features categóricas) sem algum cuidado, o desempenho da maioria dos modelos de machine learning pode ser prejudicado por tornar a base analítica muito esparsa.
Uma visão geral das classes das features numéricas em relação a target:
num_columns <- c(df %>% select_if(is.numeric) %>% colnames(), 'damaged')
df%>%
select_at(num_columns) %>%
select(-id) %>%
gather(key, value, -damaged) %>%
ggplot(aes(y=damaged, x=value))+
geom_boxplot()+
facet_wrap(~key, ncol=5, scales = "free_x")+
labs(x = "", y="")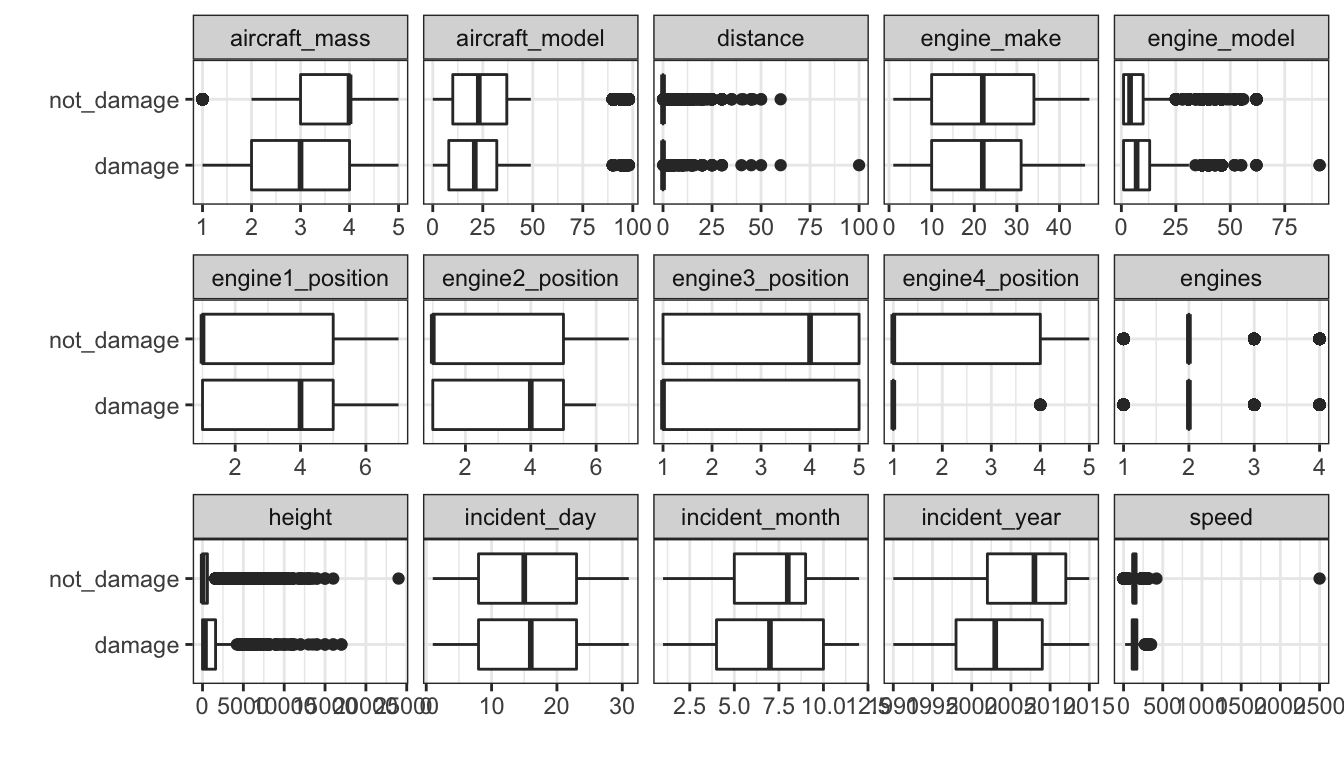
Parece que algumas features possuem comportamentos diferentes quando avaliados segundo a target. Além disso é possível notar que as features aircraft_mass, distance, engine4_position, engines, height e speed apresentam outliers.
Modelagem
Finalmente chegamos a modelagem!
Primeiro vamos definir um esquema de reamostragem (com estratificação) que será utilizado para avaliar os modelos e as métricas de qualidade.
set.seed(123)
bird_folds <- vfold_cv(df, v = 5, strata = damaged)
bird_metrics <- metric_set(mn_log_loss, accuracy, sensitivity, specificity)Nossos conjuntos de pipelines necessitarão de um pré-processador base que será comum a todos como camada inicial. Para isso utilizaremos o mesmo definido no post de referência.
base_rec <- recipe(damaged ~ ., data = df) %>%
step_select( damaged, flight_impact, precipitation,
visibility, flight_phase, engines, incident_year,
incident_month, species_id, engine_type,
aircraft_model, species_quantity, height, speed) %>%
step_novel(all_nominal_predictors()) %>%
step_other(all_nominal_predictors(), threshold = 0.01) %>%
step_unknown(all_nominal_predictors()) %>%
step_impute_median(all_numeric_predictors()) %>%
step_zv(all_predictors())Baselines
Para efeitos de comparação, vamos ajustar 2 modelos que serão utilizados como baselines para saber se a complexidade que estamos adicionando no modelo está realmente trazendo algum ganho na performance do modelo. Os modelos serão:
- Modelo nulo: um modelo que sempre prevê a classe majoritária;
- Modelo de base: Bagged Decision Tree sem adicionar pré-processamento para compensar o desequilíbrio de classe.
Modelo nulo
Avaliando modelo nulo via validação cruzada:
null_spec <- null_model(mode = "classification") %>%
set_engine("parsnip")
null_wf <-
workflow() %>%
add_recipe(base_rec) %>%
add_model(null_spec)
null_rs <-
fit_resamples(
object = null_wf,
resamples = bird_folds,
metrics = bird_metrics,
control = control_resamples(save_pred = TRUE)
)
collect_metrics(null_rs) %>% print_table(round = 5, cv = T) 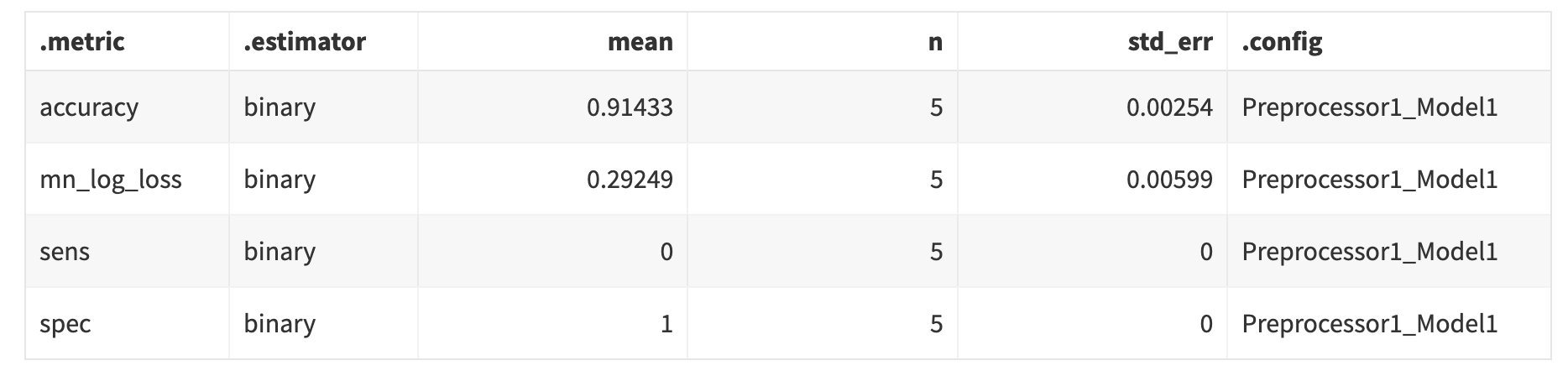
Qualquer modelo com desempenho pior do que este deve ser descartado. Vejamos a matriz de confusão:
collect_predictions(null_rs) %>%
conf_mat_plot()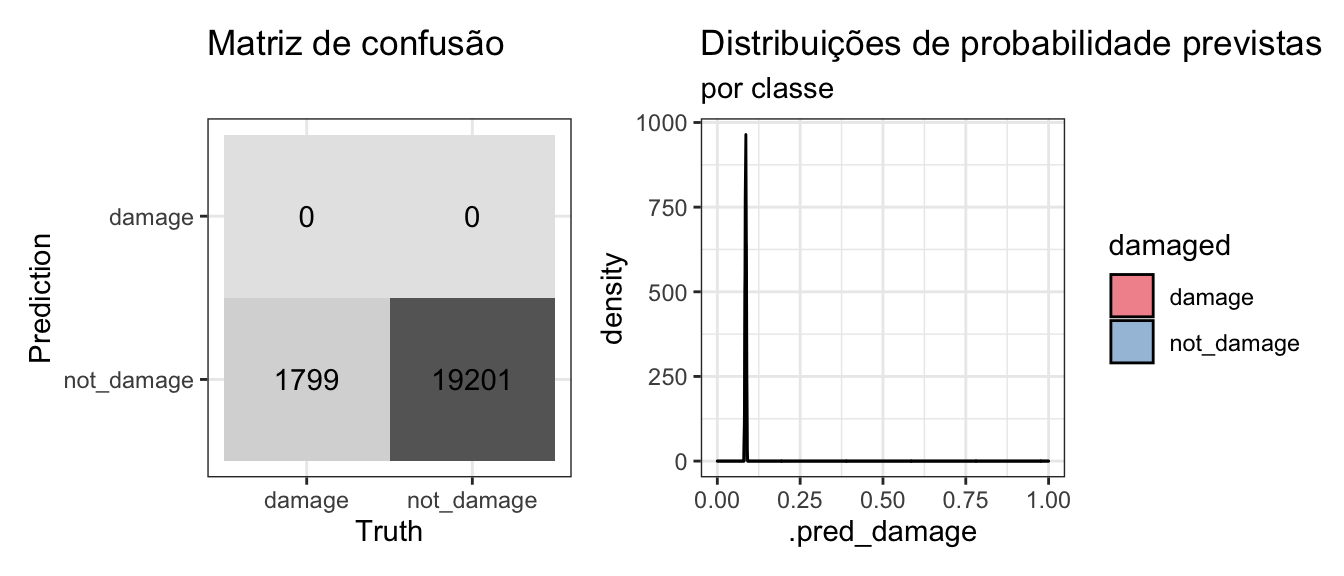
Modelo de base
Agora vamos ajusta o modelo Bagged Decision Tree sem o pré-processamento para compensar o desequilíbrio de classe:
bag_spec <-
bag_tree(min_n = 10) %>%
set_engine("rpart", times = 25) %>%
set_mode("classification")
imb_wf <-
workflow() %>%
add_recipe(base_rec) %>%
add_model(bag_spec)
set.seed(123)
imb_rs <-
fit_resamples(
imb_wf,
resamples = bird_folds,
metrics = bird_metrics,
control = control_resamples(save_pred = TRUE)
)
collect_metrics(imb_rs) %>% print_table(round = 5, cv = T)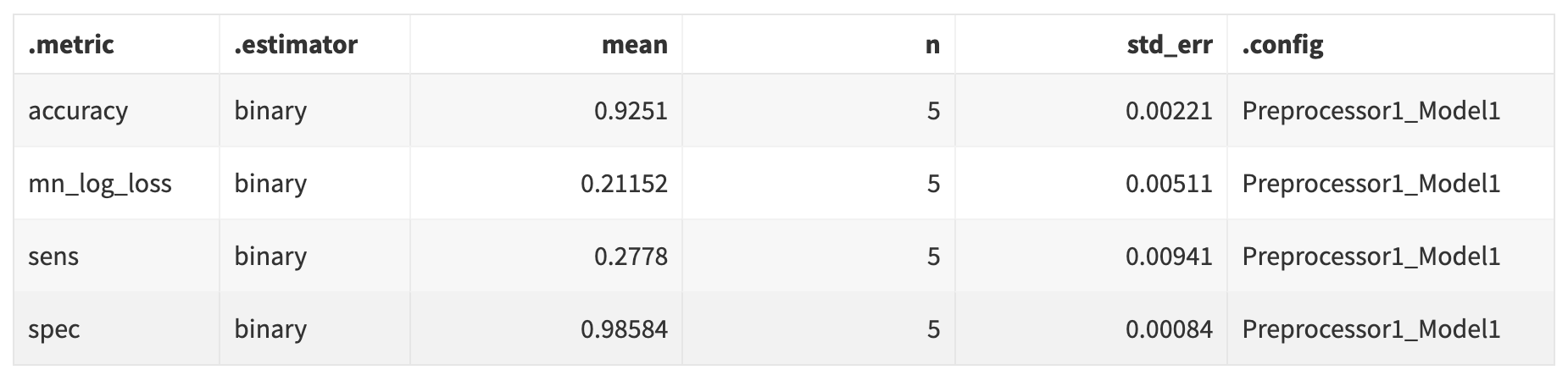
Apesar do elevado número de falsos negativos, este modelo já esta com um desempenho razoável em comparaçãao ao modelo nulo e o número de verdadeiros positivos já é quase o dobro do número de falsos positivos. Veja na matriz de confusão abaixo:
collect_predictions(imb_rs) %>%
conf_mat_plot()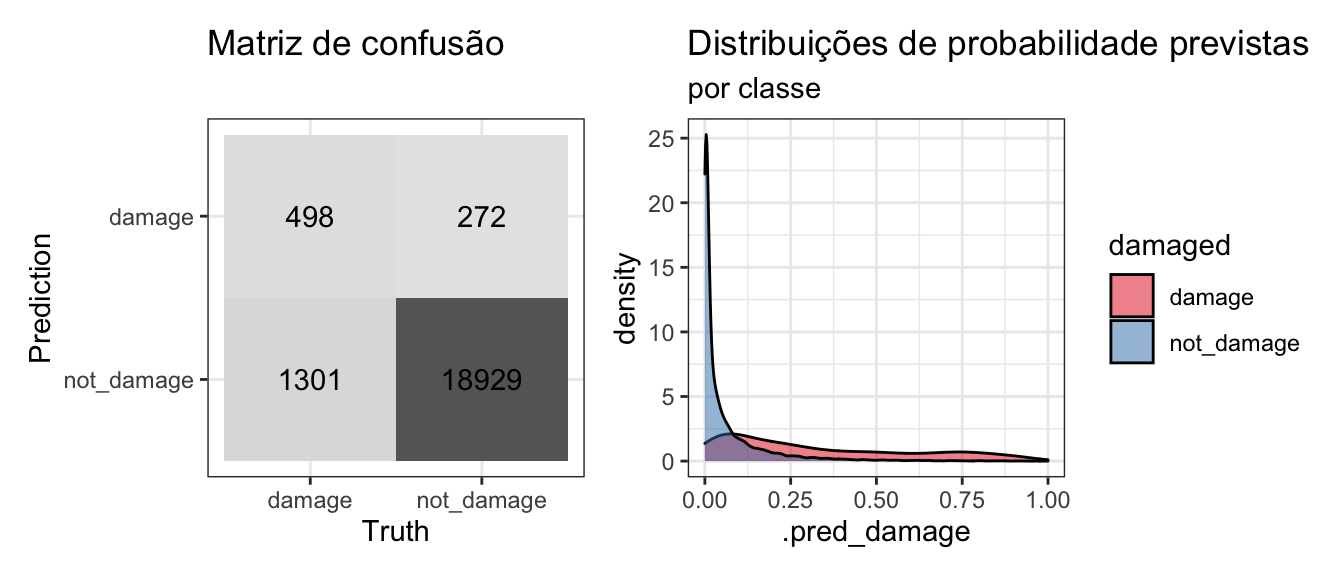
Preparar Pipeline de dados com workflowsets
A escolha do método de amostragem dos dados é tão importante quanto a escolha do modelo preditivo que será utilizado pois o desempenho pode ser enganosamente otimista visto que o algoritmo de bagging não esta usando nenhuma estratégia de subamostragem aleatória da classe majoritária em cada amostra de bootstrap para equilibrar as duas classes.
Existem muitos métodos para amostragem de dados e não há um método único que seja melhor em todos os problemas de classificação (assim como não existe o “melhor modelo”) portanto, utilizaremos este pacote para testar diferentes métodos e também tunar seus hiperparâmetros.
Oversampling
Estes métodos duplicam ou sintetizam novos dados da classe minoritária. Deve ser usado com cautela pois na vida real pode gerar alguns dados que não condizem com a relidade ou criar tantas instâncias que acaba consumindo muito mais tempo de processamento.
Random Oversampling
Este método simplesmente duplica aleatóriamente exemplos da classe minoritária. Vamos tunar esta proporção buscando números reais no intervalo [0.5,1].
rec_up <- base_rec %>%
step_upsample(damaged, over_ratio = tune())
params_up <- rec_up %>%
parameters() %>% update(over_ratio = mixture(c(0.5, 1)))SMOTE - Synthetic Minority Oversampling Technique
O SMOTE funciona gerando novos dados sintétios baseados em exemplos selecionando que estão “próximos”. Vamos tunar tanto a proporção de dados que serão gerados quanto a quantidade de vizinhos selecionados, buscando números reais e inteiros no intervalo [0.5,1] e [1, 10], respectivamente.
rec_smote <- base_rec %>%
step_dummy(all_nominal_predictors()) %>%
step_smote(damaged, over_ratio = tune(),
neighbors = tune())
params_smote <- rec_smote %>%
parameters() %>% update(over_ratio = mixture(c(0.5, 1)),
neighbors = neighbors())ADASYN - Adaptive Synthetic Sampling
O ADASYN é uma extensão do SMOTE que busca propor melhorias. Vamos tunar os mesmos parâmetros definidos no SMOTE.
rec_adasyn <- base_rec %>%
step_dummy(all_nominal_predictors()) %>%
step_adasyn(damaged,
over_ratio = tune(),
neighbors = tune())
params_adasyn <- rec_adasyn %>%
parameters() %>% update(over_ratio = mixture(c(0.5, 1)),
neighbors = neighbors())Undersampling
São técnicas que excluem ou selecionam um subconjunto de exemplos da classe majoritária e existem dezenas (se não centenas) desses métodos. Neste post utilizaremos só 3 mas existem outros implementados em outras bibliotecas (em R e em Python).
Random Undersampling
Este é o método mais simples e envolve a exclusão aleatória de algumas instâncias da classe majoritária. Vamos tunar esta proporção de frequências da minoritária para a majoritária.
rec_down <- base_rec %>%
step_downsample(damaged, under_ratio = tune())
params_down <- rec_down %>%
parameters() %>% update(under_ratio = deg_free())Near Miss Undersampling
Este algoritmo se baseia em métodos de KNN selecionando exemplos da classe majoritária que tem menor distância média dos k exemplos mais próximos. Vamos tunar tanto a proporção quanto o número de vizinhos utilizados.
rec_nearmiss <- base_rec %>%
step_dummy(all_nominal_predictors()) %>%
step_nearmiss(damaged,
under_ratio = tune(),
neighbors = tune())
params_nearmiss <- rec_nearmiss %>%
parameters() %>% update(under_ratio = deg_free(),
neighbors = neighbors())Tomek Links Undersampling
Este algoritmo que tenta excluir instâncias que sejam próximas e que possuam classes diferentes, buscando diminuir a ambiguidade dos dados. Não vamos tunar nenhum hiperparâmetro aqui.
rec_tomek <- base_rec %>%
step_dummy(all_nominal_predictors()) %>%
step_tomek(damaged)Preparar pipeline de dados
Agora que todos pipelines de dados candidatos estão definidos, vamos combinar tudo em um único objeto com workflow_set:
chi_models <-
workflow_set(
preproc = list(upsample = rec_up,
smote = rec_smote,
adasyn = rec_adasyn,
downsample = rec_down,
nearmiss = rec_nearmiss,
tomek = rec_tomek),
models = list(bag_spec = bag_spec),
cross = TRUE
)Utilizar a função option_add para adicionar as informações dos intervalos definidos para cada hiperparâmetro:
chi_models <- chi_models %>%
option_add(param_info = params_up, id = "upsample_bag_spec") %>%
option_add(param_info = params_smote, id = "smote_bag_spec") %>%
option_add(param_info = params_adasyn, id = "adasyn_bag_spec") %>%
option_add(param_info = params_down, id = "downsample_bag_spec") %>%
option_add(param_info = params_nearmiss, id = "nearmiss_bag_spec")Finalmente, vamos ajustar todos os modelos utilizando o método simples para fazer a busca dos melhores hiperparâmetros em grids de 20 valores aleatórios e calcular os scores via validação cruzada (esta parte pode demorar bastante tempo):
set.seed(123)
chi_models <-
chi_models %>%
workflow_map("tune_grid",
resamples = bird_folds,
grid = 20,
metrics = bird_metrics,
control = control_resamples(save_pred = TRUE),
verbose = TRUE)Vejamos os resultados:
rank_results(chi_models, rank_metric = "mn_log_loss", select_best = TRUE) %>%
select(-.config) %>%
mutate(wflow_id = str_remove(wflow_id, "_bag_spec")) %>%
print_table(round = 5, wf=T, height = 300, filterable = T)Matriz de confusão do modelo com menor logloss:
collect_predictions(chi_models) %>%
filter(wflow_id == "tomek_bag_spec") %>%
conf_mat_plot()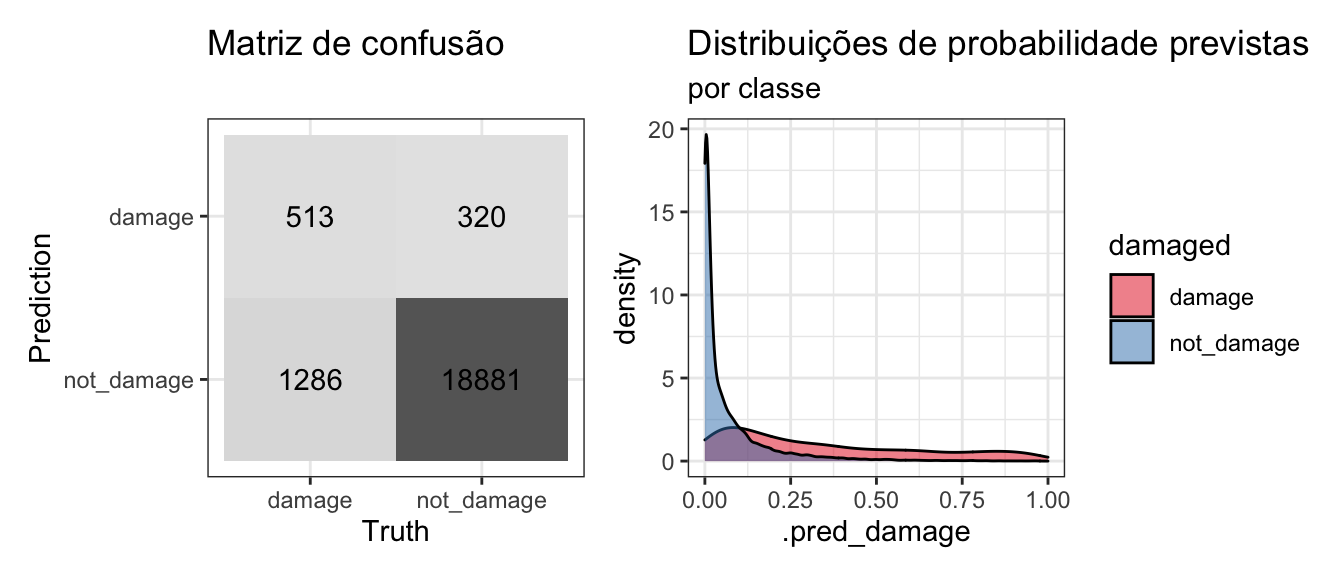
Benchmark
Comparando os resultados dos modelos ajustados:
(Clique aqui para ver o código que cria o objeto benchmark)
benchmark <- bind_rows(
mutate(collect_metrics(null_rs), wflow_id = "default", model = "null_model") %>%
select(.metric, mean, wflow_id, model) %>%
spread(.metric, mean)
,
mutate(collect_metrics(imb_rs), wflow_id = "default", model = "bag_tree") %>%
select(.metric, mean, wflow_id, model) %>%
spread(.metric, mean)
,
rank_results(chi_models, rank_metric = "mn_log_loss", select_best = TRUE) %>%
filter(wflow_id=="smote_bag_spec") %>%
select(.metric, mean, wflow_id, model) %>%
spread(.metric, mean)
,
rank_results(chi_models, rank_metric = "mn_log_loss", select_best = TRUE) %>%
filter(rank==1) %>%
select(.metric, mean, wflow_id, model) %>%
spread(.metric, mean)
) benchmark %>%
print_table(round = 5, bm = T)Como no post da Julia, a logloss e a precisão dos modelos que utilizaram métodos de balanceamento dos dados pioraram em relação ao modelo de Bagged Decision Tree sem o uso desses pipelines. Apesar da piora em relação ao modelo de base nota-se que outros métodos como Tomek Links e Adasyn se saíram ligeiramente melhores do que o Smote (além disso vimos que o Smote com sua configuração default não necessariamente produrizá os melhores resultados).
Este tipo de performance é muito comum e até esperado visto que estamos avaliando o modelo através de uma única métrica (com os mesmos pontos de corte e com o mesmo algoritmo). Normalmente no mundo real monitoramos diversas métricas e experimentamos mais configurações de hiperparâmetros de diferentes modelos com diferentes pipelines.
Conclusão
Assim como não existe melhor modelo, não existe melhor técnica de balanceamento de dados. Portanto, na busca de melhores resultados nós podemos tentar otimizar qual abordagem será uyilizada bem como seus hiperparâmetros (em conjunto com os hiperparâmetros dos modelos em questão).
Esta abordagem em R é nova para mim (estou mais acostumado a utilizar em Python com o método sklearn.pipeline.Pipeline em conjunto com a biblioteca imblearn) então qualquer crítica e sugestão de melhoria será muito bem vinda! Basta entrar em contato ou deixar aqui nos comentários!
Bons estudos e espero que gostem! 🚀
Referências
- https://www.tidyverse.org/blog/2021/03/workflowsets-0-0-1/
- https://www.kaggle.com/c/sliced-s01e02-xunyc5
- https://juliasilge.com/blog/sliced-aircraft/
- https://topepo.github.io/caret/subsampling-for-class-imbalances.html
- https://machinelearningmastery.com/bagging-and-random-forest-for-imbalanced-classification/
- https://machinelearningmastery.com/what-is-imbalanced-classification/
- https://machinelearningmastery.com/framework-for-imbalanced-classification-projects/
- https://machinelearningmastery.com/data-sampling-methods-for-imbalanced-classification/






























Share this post
Twitter
LinkedIn
Email