Pacotes do R para avaliar o ajuste de modelos
Alguns pacotes úteis para avaliar o ajuste do modelo de forma rápida, precisa e elegante

Funções do R para avaliar o ajuste de modelos
Traduzindo:
“Essencialmente, todos os modelos estão errados, mas alguns são úteis” - George E. P. Box
Se você estuda estatística provavelmente já deve saber quem é este simpático senhor. Box teve grande contribuição para a estatística. Foi aluno do Ronald Aylmer Fisher e ainda se casou com a filha dele!
Lendo um artigo sobre a vida de Fisher um parágrafo me chamou atenção com uma fala de sua filha, que dizia o seguinte:
“Joan Fisher Box, filha de Fisher, em seu livro sobre a vida do pai, se referindo à péssima classificação dele em francês, escreveu: “… ele nunca teve muita paciência com irrelevâncias.” (Box, 1978)"
Fico imaginando o tamanho da contribuição desdes crânios para a comunidade se tivessem acesso a tantos mecanismos que temos hoje em dia e o que eles achariam relevantes..
Para o bom ajuste de um modelo, certamente; a inferência, as análises de desvios, os critérios de seleção de um modelo, conferir comportamento dos resíduos e avaliação das estatísticas de diagnósticos são muito relevantes.
No CRAN já contamos com muitos pacotes disponíveis para nos auxiliar nessas avaliações, portanto vou mostrar aqui alguns pacotes com funções que já me ajudaram muito em avaliações de modelos indo além das funções nativas do R e do pacote ggplot2 (Um excelente pacote para apresentações elegantes e práticas de resultados visuais).
GGally
Este pacote é sensacional, existem funções muito relevantes nele para melhorar a nossa experiência com ajuste de modelos, as funções apresentadas aqui são baseadas na página de documentação GGally, lá você pode conferir a documentação completa.
Primeiramente vamos carregar o pacote:
library(GGally)Carregado o pacote, vejamos as principais funções que podem nos auxiliar.
GGally::ggcoef
O objetivo da função GGally::ggcoef é traçar rapidamente os coeficientes de um modelo.
Para um modelo linear:
reg <- lm(Sepal.Length ~ Sepal.Width + Petal.Length + Petal.Width, data = iris)
ggcoef(reg)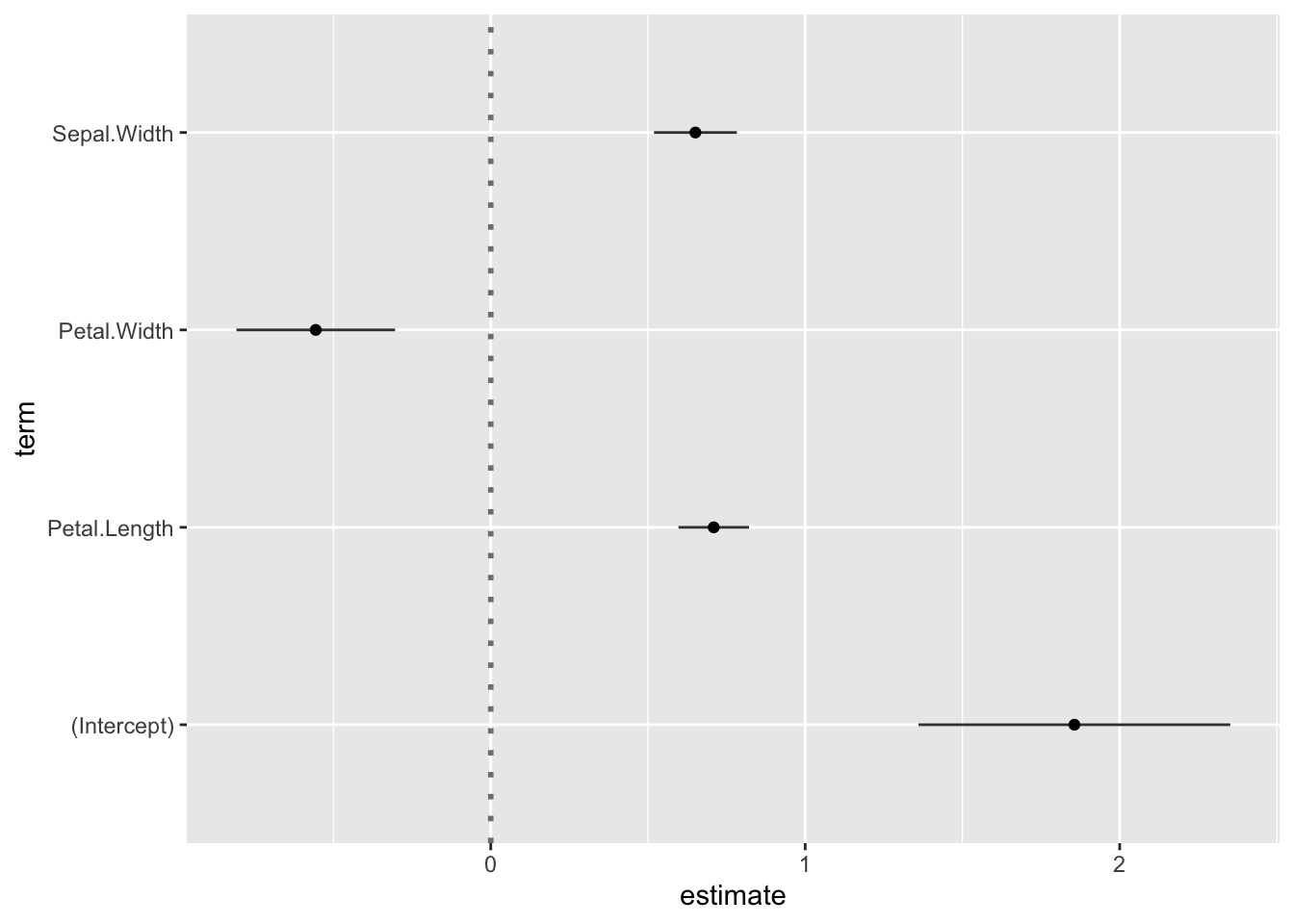
Para um modelo logístico podemos utilizar o argumento exponentiate = TRUE e além disso, somos capazes de fazer diversas alterações no gráfico utilizando o ggcoef() veja alguns exemplo de argumentos que podem ser usados para personalizar como barras de erro e a linha vertical são plotadas:
#Ajustando o modelo:
d <- as.data.frame(Titanic)
log.reg <- glm(Survived ~ Sex + Age + Class, family = binomial, data = d, weights = d$Freq)
#Elaborando o gráfico
ggcoef(
log.reg, #O modelo a ser conferido
exponentiate = TRUE, #Para avaliar o modelo logístico
vline_color = "red", #Reta em zero
#vline_linetype = "solid", #Altera a linha de referência
errorbar_color = "blue", #Cor da barra de erros
errorbar_height = .25,
shape = 18, #Altera o formato dos pontos centrais
#size=3, #Altera o tamanho do ponto
color="black", #Altera a cor do ponto
mapping = aes(x = estimate, y = term, size = p.value))+
scale_size_continuous(trans = "reverse") #Essa linha faz com que inverta o tamanho 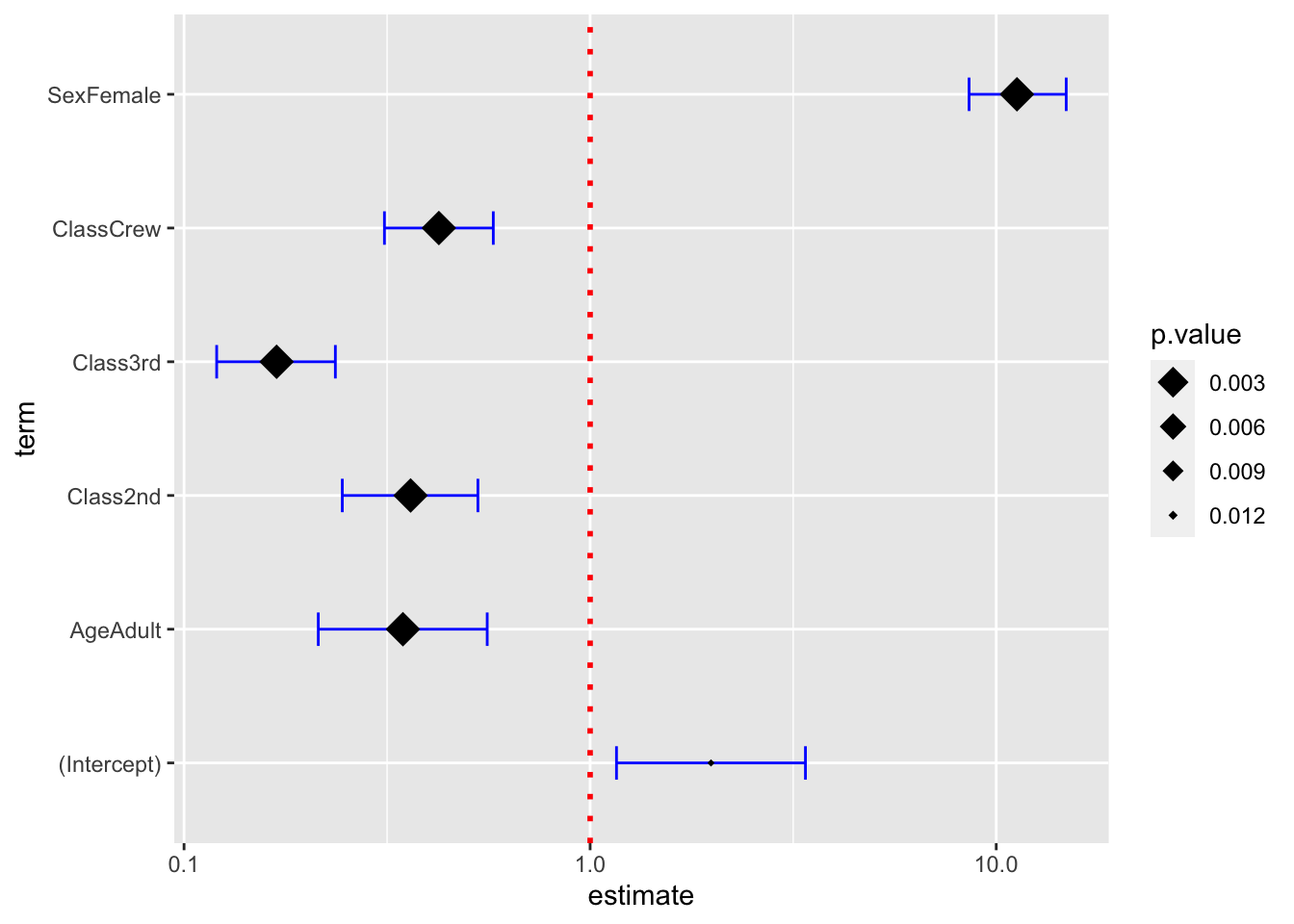
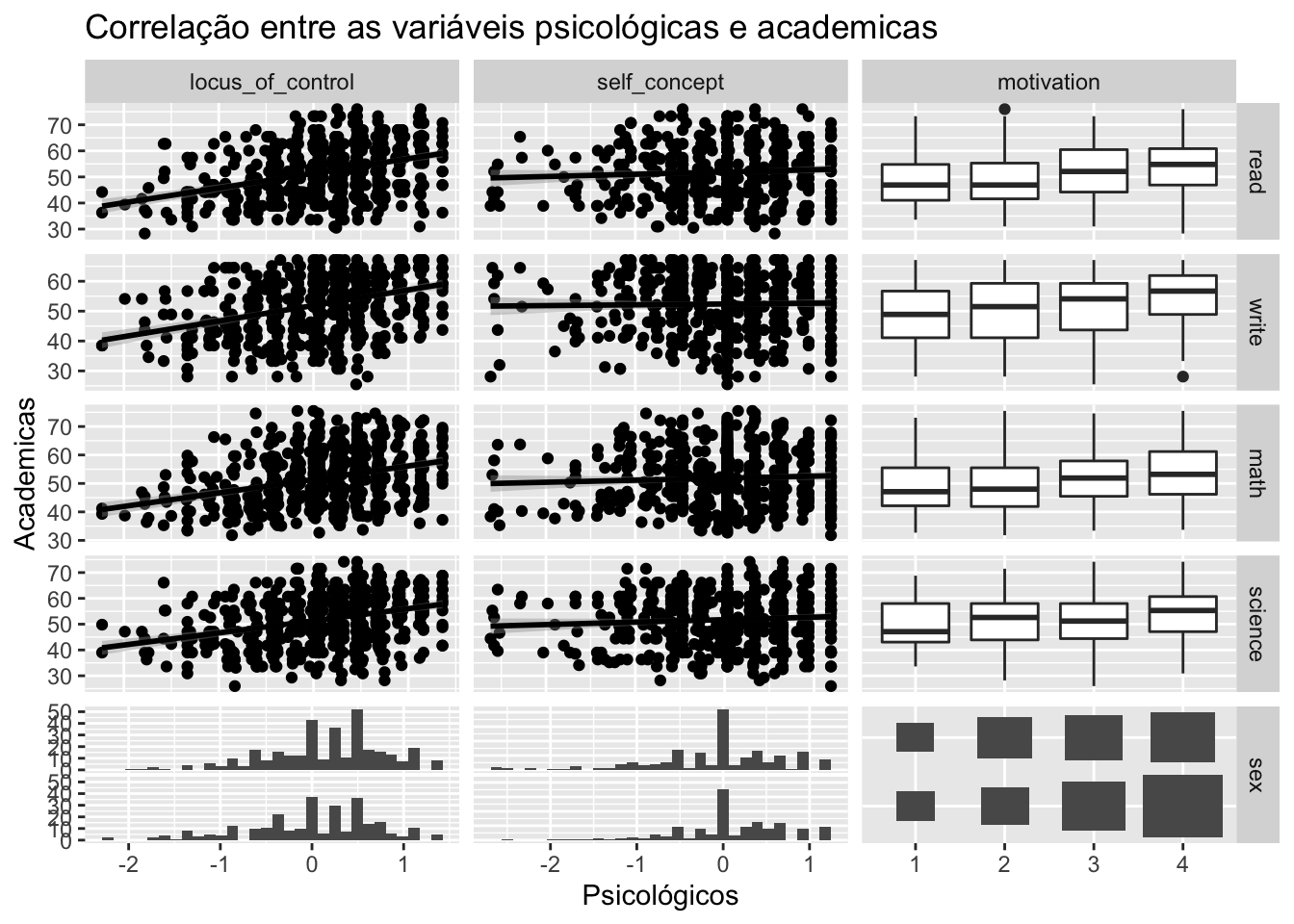
GGally::ggduo
O objetivo desta função é exibir dois dados agrupados em uma matriz de plotagem. Isso é útil para análise de correlação canônica, análise de séries temporais múltiplas e análise de regressão.
Os dados do exemplo apresentados aqui podem ser encontrados neste link
data(psychademic)
head(psychademic)## locus_of_control self_concept motivation read write math science sex
## 1 -0.84 -0.24 4 54.8 64.5 44.5 52.6 female
## 2 -0.38 -0.47 3 62.7 43.7 44.7 52.6 female
## 3 0.89 0.59 3 60.6 56.7 70.5 58.0 male
## 4 0.71 0.28 3 62.7 56.7 54.7 58.0 male
## 5 -0.64 0.03 4 41.6 46.3 38.4 36.3 female
## 6 1.11 0.90 2 62.7 64.5 61.4 58.0 femalepsych_variables <- attr(psychademic, "psychology")
academic_variables <- attr(psychademic, "academic")ggduo(
psychademic, psych_variables, academic_variables,
types = list(continuous = "smooth_lm"),
title = "Correlação entre as variáveis psicológicas e academicas",
xlab = "Psicológicos",
ylab = "Academicas"
)
Uma vez que o ggduo não tem uma seção superior para exibir os valores de correlação, podemos usar uma função personalizada para adicionar a informação nas parcelas contínuas.
Criando uma função personalizada para informar a correlação entre as observações:
lm_with_cor <- function(data, mapping, ..., method = "pearson") {
x <- eval(mapping$x, data)
y <- eval(mapping$y, data)
cor <- cor(x, y, method = method)
ggally_smooth_lm(data, mapping, ...) +
ggplot2::geom_label(
data = data.frame(
x = min(x, na.rm = TRUE),
y = max(y, na.rm = TRUE),
lab = round(cor, digits = 3)
),
mapping = ggplot2::aes(x = x, y = y, label = lab),
hjust = 0, vjust = 1,
size = 5, fontface = "bold",
inherit.aes = FALSE # do not inherit anything from the ...
)
}Portanto:
ggduo(
psychademic, psych_variables, academic_variables,
types = list(continuous = "smooth_lm"),
title = "Correlação entre variáveis acadêmica e psicológica",
xlab = "Psicológica",
ylab = "Academica"
)+
theme(legend.position = "bottom")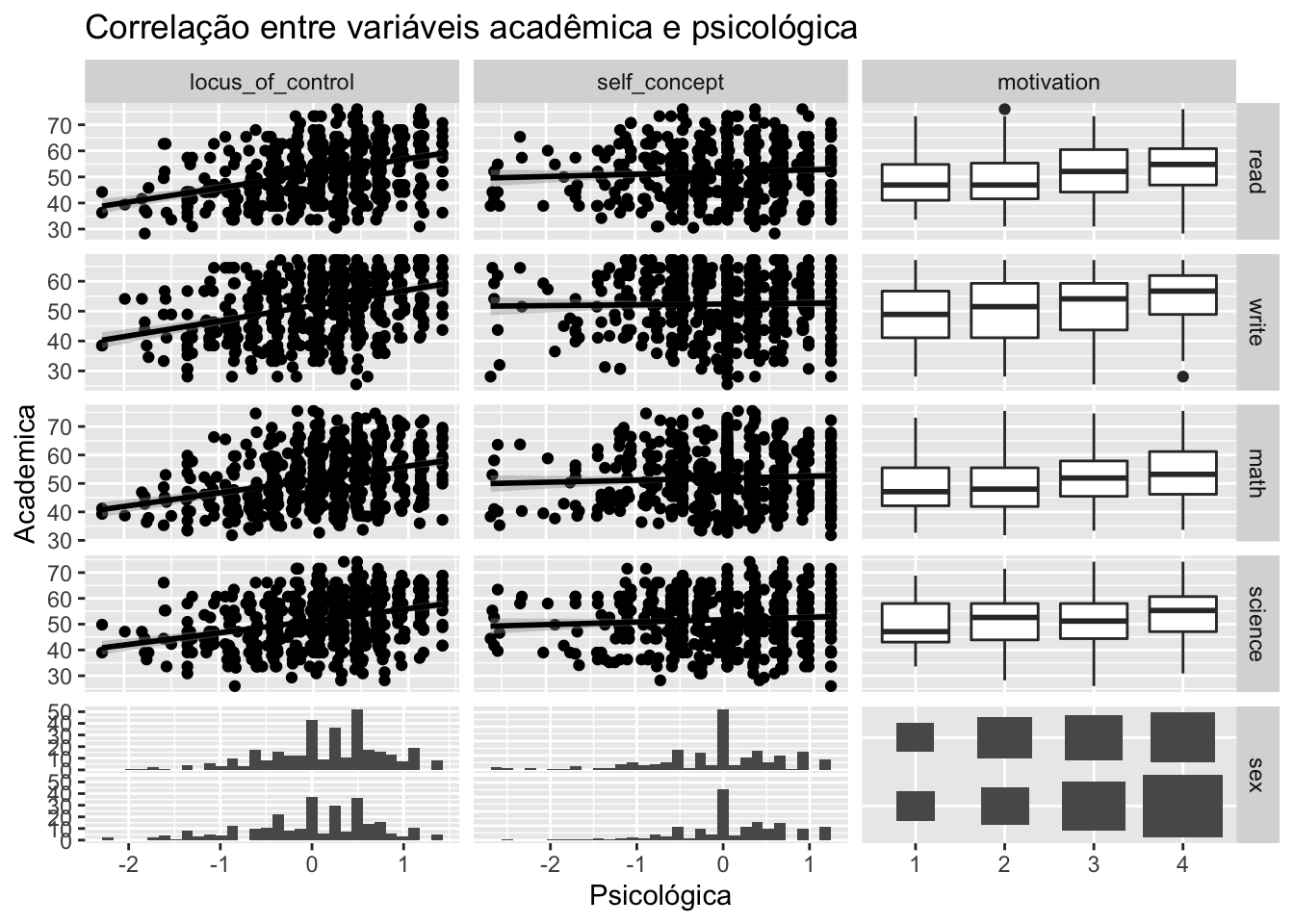
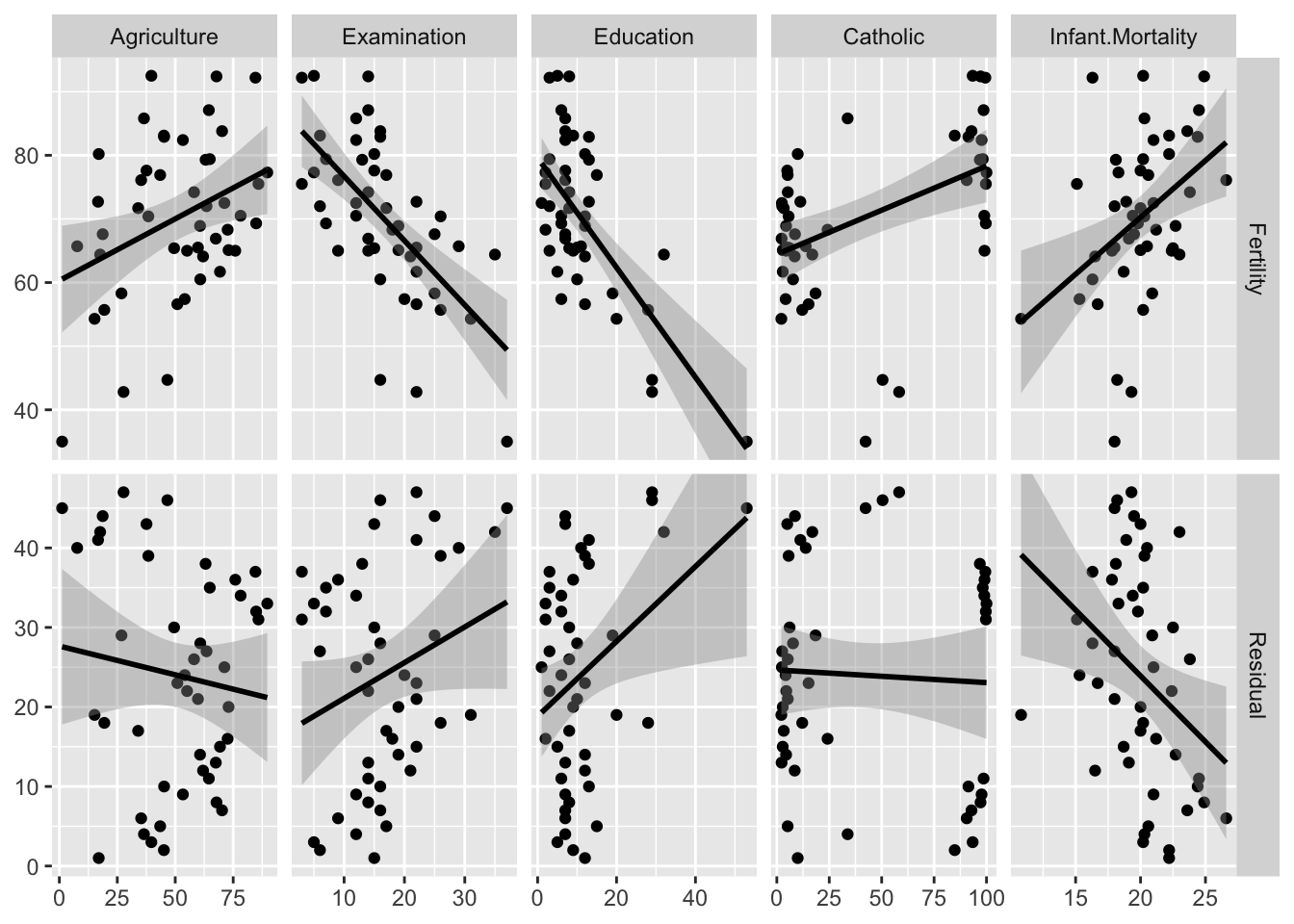
Para avaliar resíduos da uma regressão ajustada para cada uma das variáveis explanatórias vs. as variáveis explanatórias:
dados <- datasets::swiss
# Criando uma coluna "fake":
dados$Residual <- seq_len(nrow(dados))
# Calculando todos os resíduos que serão exibidos:
colunas=2:6 #Informe as colunas que contem as variaveis explanatorias
residuals <- lapply(dados[colunas], function(x) {
summary(lm(Fertility ~ x, data = dados))$residuals
})
# Calculando um intervalo constante para todos os resíduos
y_range <- range(unlist(residuals))
# Função modificada para mostrar os resíduos:
lm_or_resid <- function(data, mapping, ..., line_color = "red", line_size = 1) {
if (as.character(mapping$y) != "Residual") {
return(ggally_smooth_lm(data, mapping, ...))
}
# Criando os resíduos para apresentar:
resid_data <- data.frame(
x = data[[as.character(mapping$x)]],
y = residuals[[as.character(mapping$x)]]
)
ggplot(data = data, mapping = mapping) +
geom_hline(yintercept = 0, color = line_color, size = line_size) +
ylim(y_range) +
geom_point(data = resid_data, mapping = aes(x = x, y = y), ...)
}
# Plote os dados:
ggduo(
dados,
2:6, c(1,7),
types = list(continuous = lm_or_resid)
)
GGally::ggnostic
O ggnostic é um wrapper de exibição para ggduo que exibe diagnósticos de modelo completo para cada variável explicativa dada.
Por padrão, o ggduo exibe os valores residuais, o sigma do modelo de “leave-one-out”, os pontos de alavanca e a distância de Cook em relação a cada variável explicativa.
As linhas da matriz de plotagem podem ser expandidas para incluir valores ajustados, erro padrão dos valores ajustados, resíduos padronizados e qualquer uma das variáveis de resposta.
Se o modelo for um modelo linear, os asteriscos (*) são adicionados de acordo com a significância anova de cada variável explicativa.
A maioria das parcelas diagnósticas contêm linhas de referência para ajudar a determinar se o modelo está adequadamente instalado
Olhando para os conjuntos de dados do conjunto de dados state.x77 ajustaremos um modelo de regressão múltipla para a expectativa de vida.
#Dados que serão utilizados no exemplos:
state <- as.data.frame(state.x77)
#Arrumando o nome das variaveis:
colnames(state)[c(4, 6)] <- c("Life.Exp", "HS.Grad")
# Ajustando o modelo completo:
model <- lm(Life.Exp ~ ., data = state)
# Executando o stepwise para encontrar o melhor ajuste
model <- step(model, trace = FALSE)Executando o diagnóstico deste modelo com a função ggnostic():
# look at model diagnostics
ggnostic(model)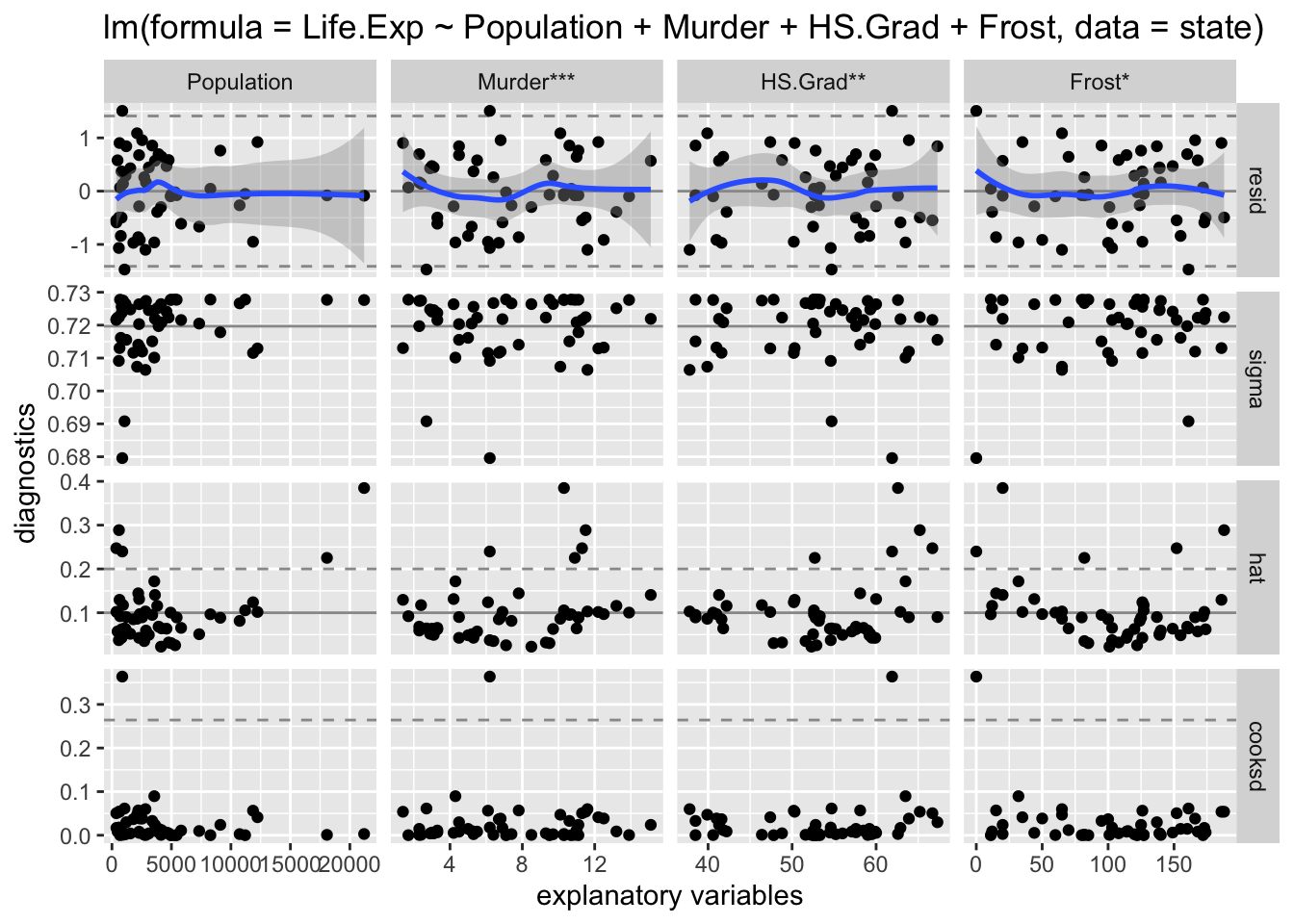
Para acessar as variáveis influentes do modelo podemos utilizar a função influence.measures(), veja:
summary(influence.measures(model))## Potentially influential observations of
## lm(formula = Life.Exp ~ Population + Murder + HS.Grad + Frost, data = state) :
##
## dfb.1_ dfb.Pplt dfb.Mrdr dfb.HS.G dfb.Frst dffit cov.r cook.d
## Alaska 0.41 0.18 -0.40 -0.35 -0.16 -0.50 1.36_* 0.05
## California 0.04 -0.09 0.00 -0.04 0.03 -0.12 1.81_* 0.00
## Hawaii -0.03 -0.57 -0.28 0.66 -1.24_* 1.43_* 0.74 0.36
## Nevada 0.40 0.14 -0.42 -0.29 -0.28 -0.52 1.46_* 0.05
## New York 0.01 -0.06 0.00 0.00 -0.01 -0.07 1.44_* 0.00
## hat
## Alaska 0.25
## California 0.38_*
## Hawaii 0.24
## Nevada 0.29
## New York 0.23Esta função retorna as seguintes estatísticas:
| DFBeta | DFFit | CovRatio | D.Cook | h |
|---|---|---|---|---|
| Alteração no vetor estimado \(\hat \beta\) ao se retirar o i-ésimo ponto da análise | Alteração provocada no valor ajustado pela retirada da observação \(i\) | Expressa o relação de covariancia | Medida de afastamento das estimativas ao retirar \(i\) e também considera o resíduo estudentizado internamente | Elementos da diagonal da matriz H |
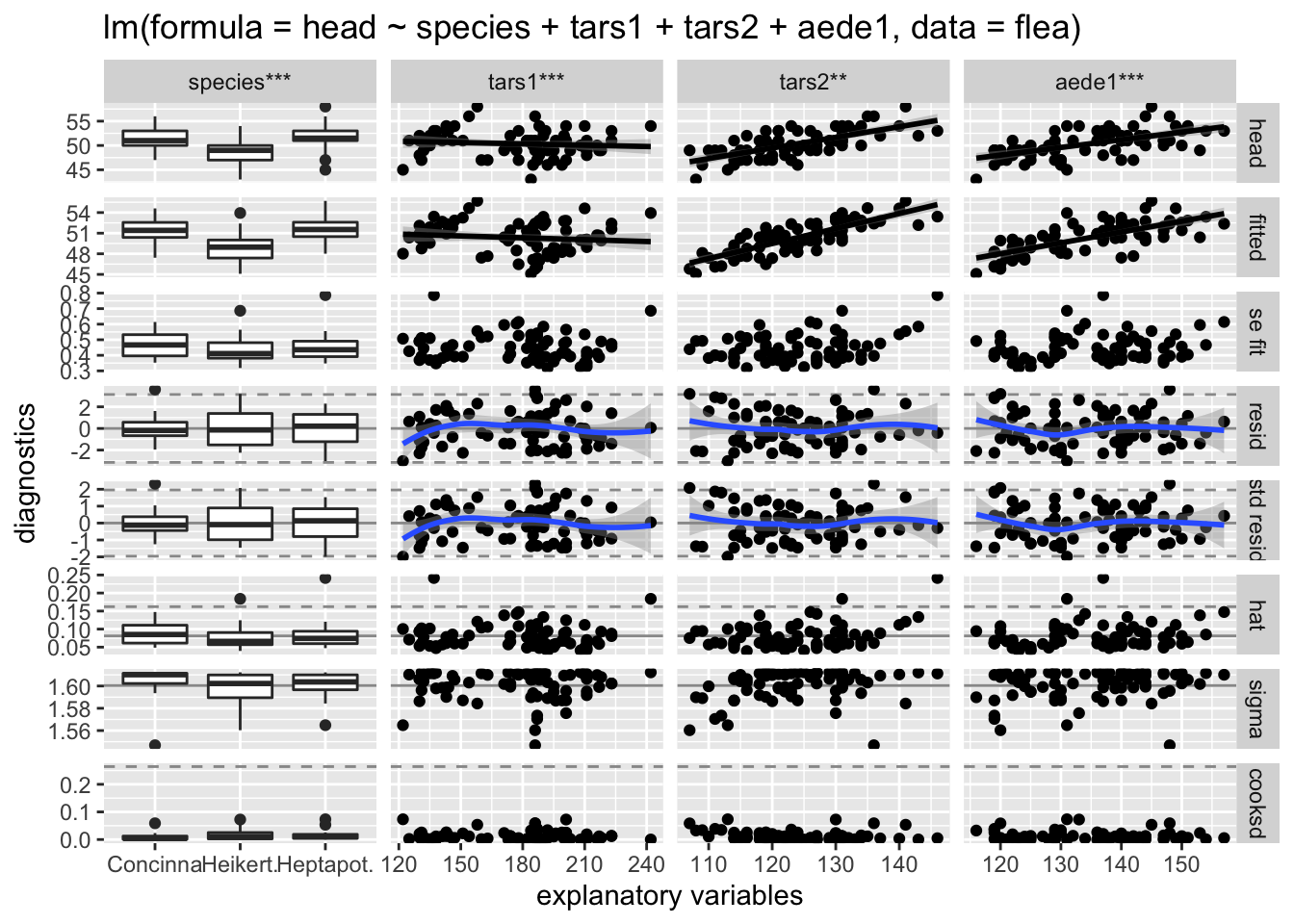
Vejamos então um exemplo de matriz de matriz de diagnóstico completo.
As seguintes linhas de código exibirão uma matriz de diagnóstico para o mesmo modelo:
#Ajustando um modelo de exemplo:
flea_model <- step(lm(head ~ ., data = flea), trace = FALSE)Todas as colunas possíveis e usando ggally_smooth() para exibir os pontos ajustados e as variáveis de resposta temos:
# default output
ggnostic(flea_model,
# mapping = ggplot2::aes(color = species), #Para colorir segundo um fator
columnsY = c("head", ".fitted", ".se.fit", ".resid", ".std.resid", ".hat", ".sigma", ".cooksd"),
continuous = list(default = ggally_smooth, .fitted = ggally_smooth)
)
GGally::ggpairs
O ggpairs é uma forma especial de uma ggmatrix que produz uma comparação pairwise de dados multivariados. Por padrão, o ggpairs fornece duas comparações diferentes de cada par de colunas e exibe a densidade ou a contagem da variável respectiva ao longo da diagonal. Com diferentes configurações de parâmetros, a diagonal pode ser substituída pelos valores do eixo e rótulos variáveis.
#Funcao de correlacoes
my_fn <- function(data, mapping, method="lm", ...){
p <- ggplot(data = data, mapping = mapping) +
geom_point() +
geom_smooth(method=method, ...)
p
}
data(tips, package = "reshape")
#Correlaçoes cruzadas
ggpairs(tips, lower = list(continuous = my_fn))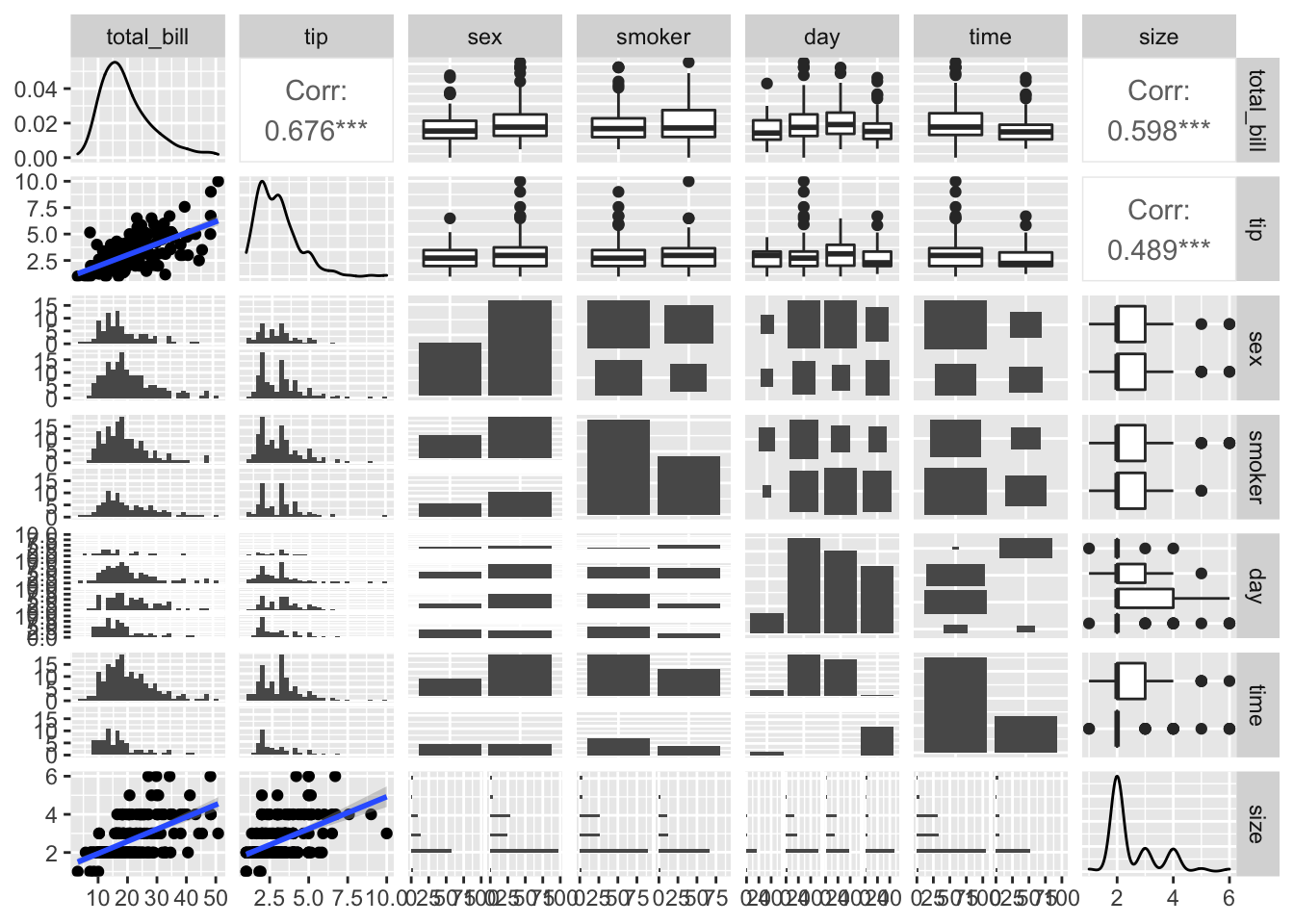
Existem muitos recursos ocultos dentro dos ggpairs() e muitos exemplos podem ser conferidos na internet para obter o máximo do ggpairs().
GGally::ggscatmat
A principal função é ggscatmat. É semelhante a ggpairs(), mas funciona apenas para dados multivariados puramente numéricos.
É mais rápido que ggpairs, porque é necessário fazer menos escolhas.
Ele cria uma matriz com diagramas de dispersão na diagonal inferior, densidades na diagonal e correlações escritas na diagonal superior.
A sintaxe é inserir o conjunto de dados, as colunas que deseja traçar, uma coluna de cores e um nível alfa.
data(flea)
ggscatmat(flea, columns = 2:4, color="species", alpha=0.8)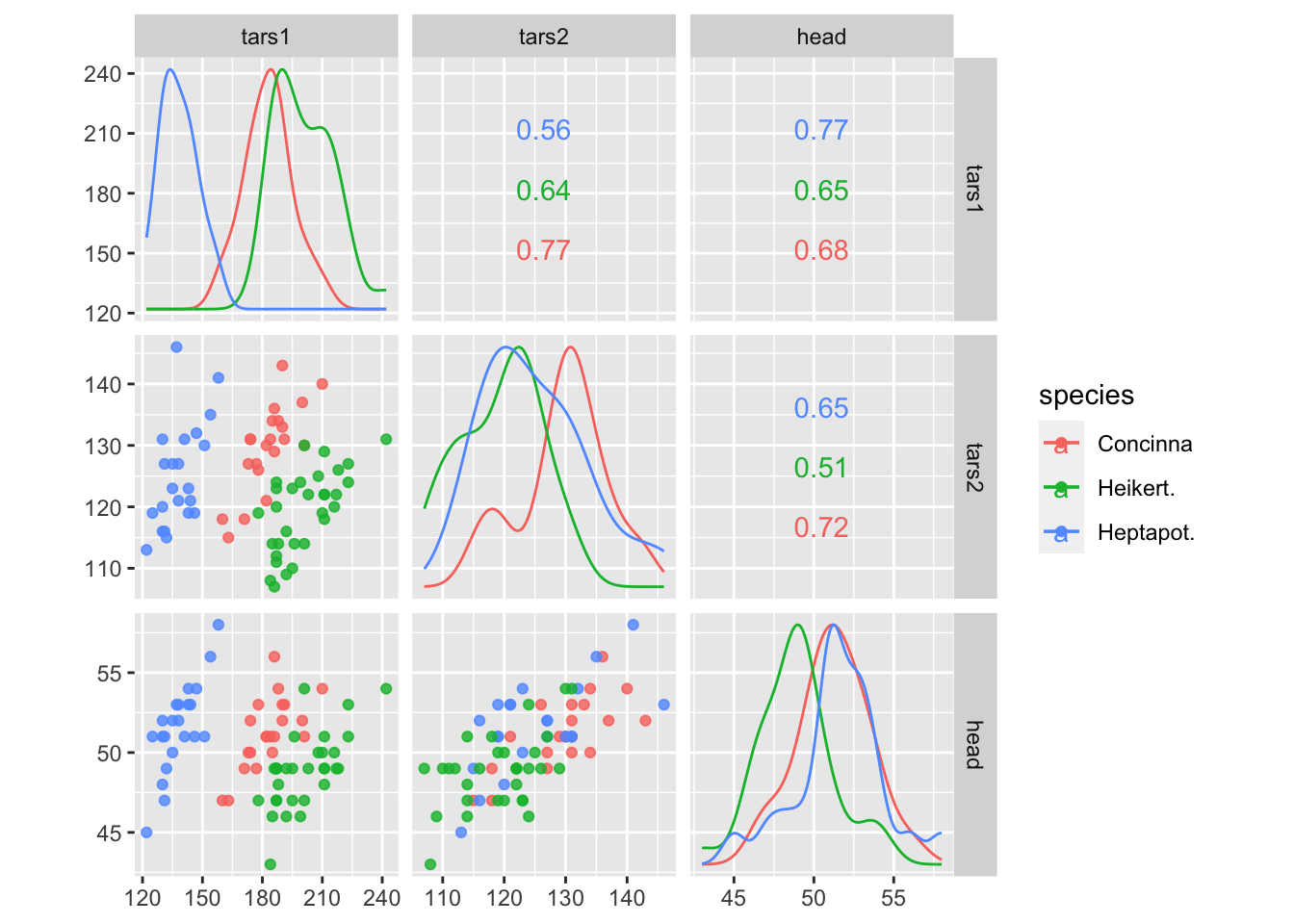
ggfottify
Outra opção interessante para avaliar o ajuste dos modelos é o pacote ggfottify. Ele disponibiliza uma interface de traçado (como a função plot(modelo_ajustado)) de análise e gráficos em um estilo unificado, porém usando ggplot2.
Vamos então dar início carregando o pacote:
library(ggfortify)Veja a seguir alguns dos gráficos disponíveis no R para a análise de resíduos:
autoplot(flea_model, which = 1:6, ncol = 3, label.size = 3)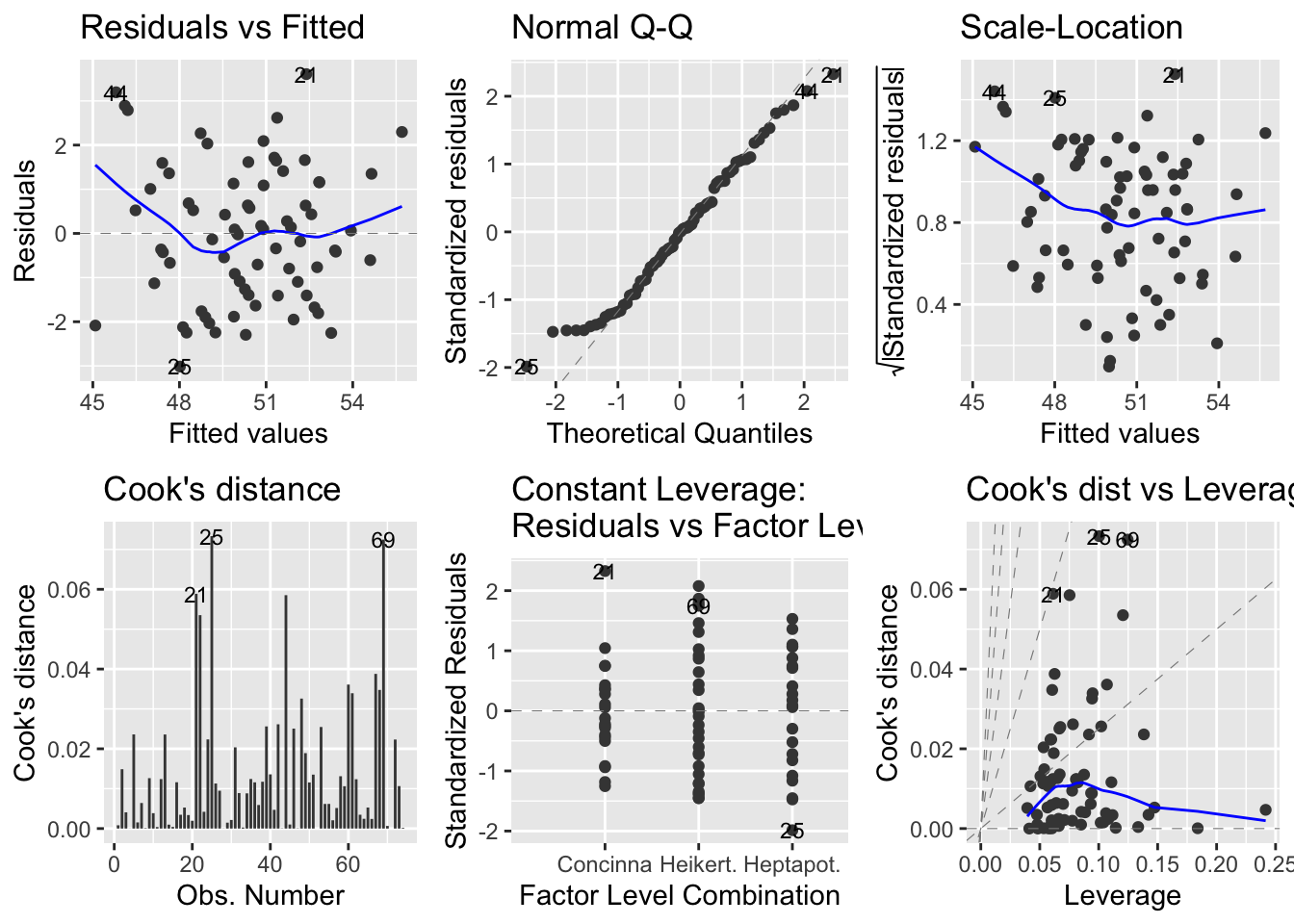
Especificando as opções de plot
Algumas propriedades desses gráficos podem ser alteradas. Por exemplo, a opção colour = 'dodgerblue3' é para pontos de dados, o smooth.colour = 'black' é para linhas de suavização e ad.colour = 'blue' é para opções adicionais.
Veja ainda que ncol e nrow controlam o layout.
autoplot(flea_model, which = 1:6, colour = 'dodgerblue3',
smooth.colour = 'black', smooth.linetype = 'dashed',
ad.colour = 'blue',
label.size = 3, label.n = 5, label.colour = 'blue',
ncol = 3)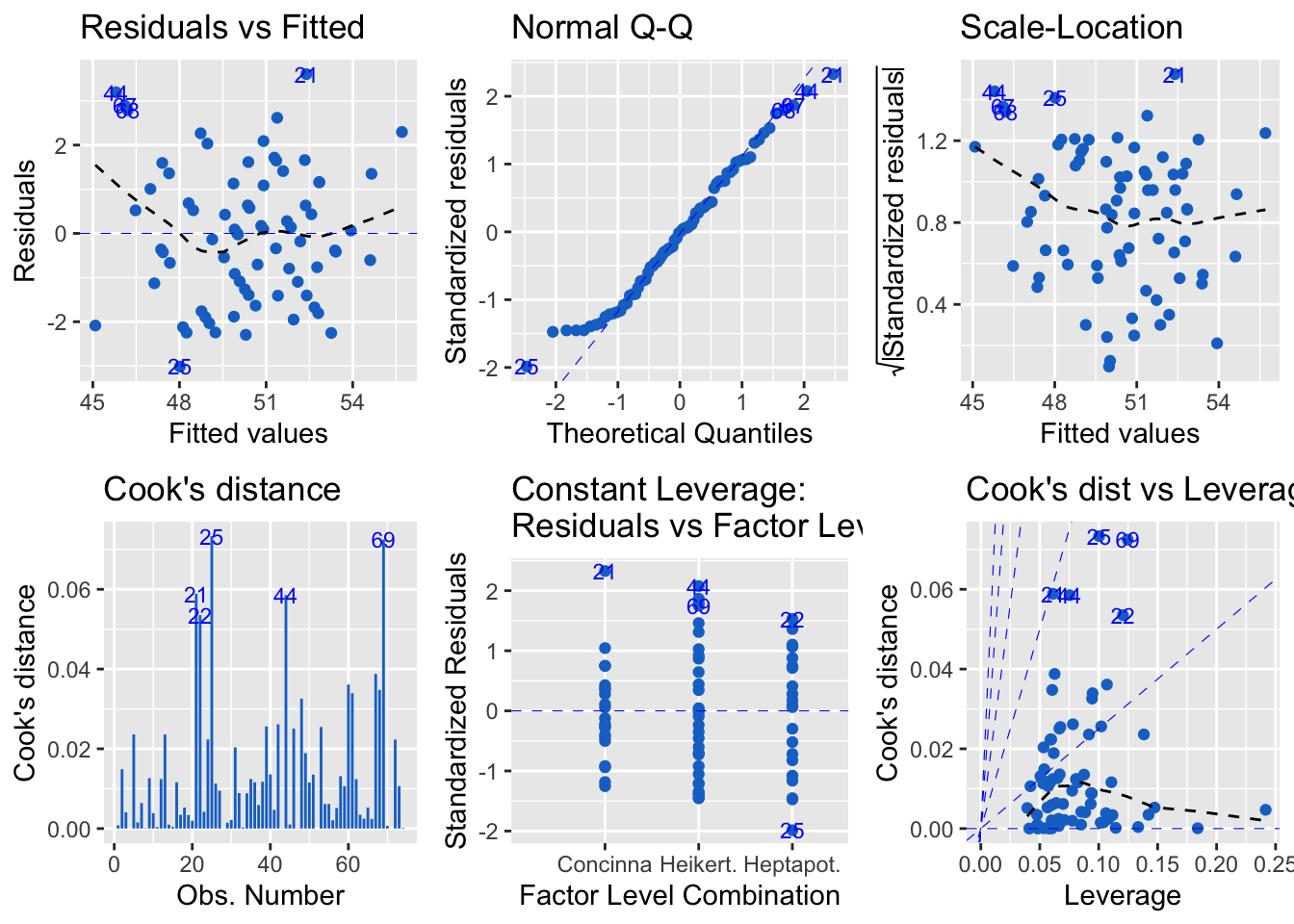
Além disso, você pode usar nomes de colunas para essas propriedades, vamos separar os grupos de machos e fêmeas por cores:
autoplot(flea_model, which = 1:6, data = flea,
colour = 'species', label.size = 3,
ncol = 3)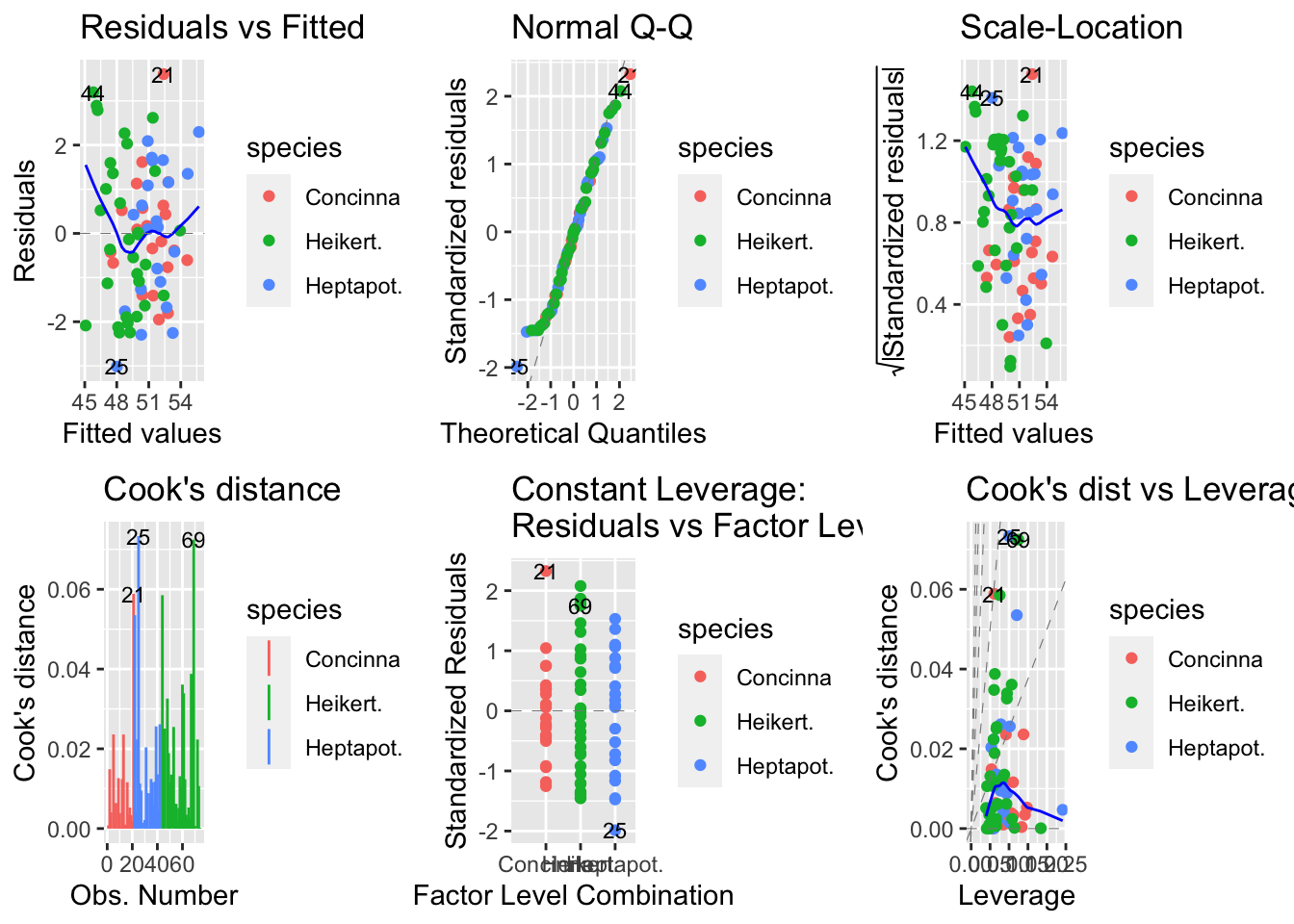
O que será que os crânios da estatística fariam diante de tantos recursos?




















Share this post
Twitter
LinkedIn
Email