Hackeando o R: estratégia split-apply-combine
Veja como aplicar essa estratégia de maneira eficiente utilizando os pacotes do tidyverse: dplyr+tidyr+purrr
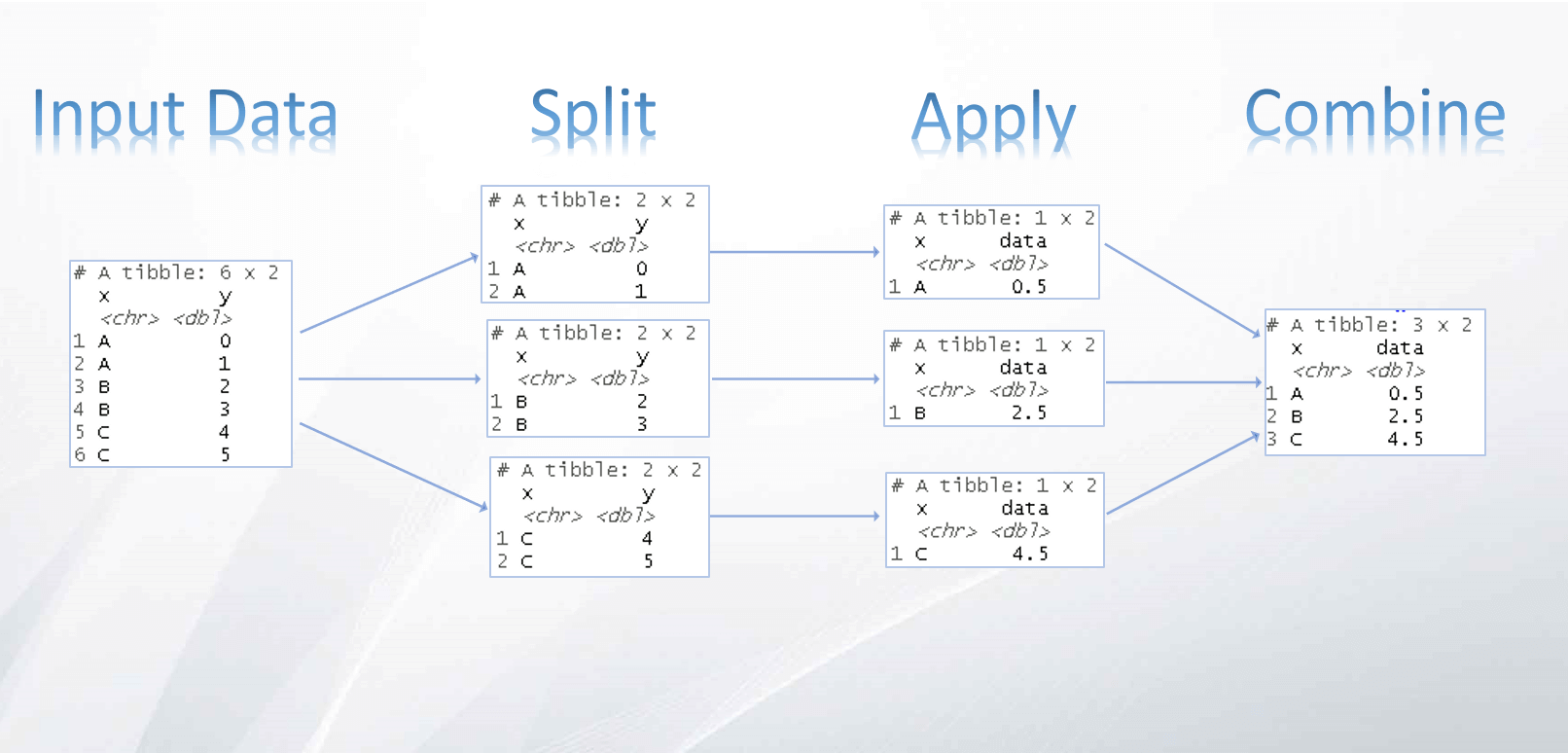
O método split-apply-combine
Geralmente em uma análise de dados precisamos compreender, além do comportamento geral dos dados, o seu comportamento de acordo com alguns segmentos.
No famoso paper The Split-Apply-Combine Strategy for Data Analysis, Hadley Wickham descreve a abordagem “split-apply-combine” (dividir-aplicar-combinar) como uma das mais comuns em uma análise de dados. Em R essa tarefa pode ser feita por diversos caminhos, veja alguns dos modos de se fazer utilizando funções base do R e abordagens mais antigas:
split()+lapply()+do.call(rbind, ...)ddply()do pacoteplyrgroup_by+do()split()+map_dfr()
Todos esses exemplos atendem à maioria dos casos que deseja-se utilizar a abordagem “split-apply-combine”, porém, veja por exemplo este tópico na community.rstudio.com criado no final de 2017 em que ocorre um comunicado que a função do() será descontinuada
Ou ainda, confira quando foi o último lançamento de atualização do pacote plyr no CRAN (foi em junho de 2016).
Com a proposta de mais eficiência e legibilidade do código, atualmente existem maneiras mais sofisticadas e modernas de se realizar esta tarefa com pacotes que foram atualizados já este ano de 2019. Veja nas seções a seguir o aumento de produtividade que é possível se obter combinando os pacotes dplyr, tidyr e purrr da coleção de pacotes do tidyverse.
Usando só o dplyr
Usamos “split-apply-combine” implicitamente o tempo todo quando utilizamos as funções groupy_by() + summarise() do pacote dplyr
Poderíamos facilmente reproduzir o exemplo da imagem do post com os seguintes comandos:
library(dplyr)
data <- tibble(x = c("A", "A", "B", "B", "C", "C"),
y = c(0,1,2,3,4,5))
data %>% # input data
group_by(x) %>% # split
summarise(data = mean(y)) # apply/combine## # A tibble: 3 x 2
## x data
## <chr> <dbl>
## 1 A 0.5
## 2 B 2.5
## 3 C 4.5Essa sequência de códigos aplica a abordagem implicitamente, agrupando os dados de acordo com a variável selecionada e em seguida aplicando a operação e combinando os resultados em uma matriz resumida
Usando dplyr + tidyr + purrr
Poderíamos ter realizado a mesma operação de forma explícita com o auxílio das funções nest(), map(), mutate() e unnest() dos pacotes dplyr tidyr e purrr, veja:
# Pacotes necessários
library(tidyr)
library(purrr)
# Dados
data <- tibble(x = c("A", "A", "B", "B", "C", "C"),
y = c(0,1,2,3,4,5))
# Codigos
data %>% # Input Data
nest(-x) %>% # Split
mutate(data = map(data, ~mean(.x$y))) %>% # Apply
unnest() # Combine## # A tibble: 3 x 2
## x data
## <chr> <dbl>
## 1 A 0.5
## 2 B 2.5
## 3 C 4.5Note que obtemos a mesma saída do código anterior
Split-Apply-Combine com funções complexas
Você deve estar se perguntando:
“Tá, eu tenho um atalho para usar a estratégia ”split-apply-combine” com pacote dplyr, por que eu preciso usar os dados aninhados?”
Trabalhar com dados aninhados permite aplicar qualquer tipo de função em partições do conjunto de dados e juntar os resultados em um objeto do tipo tibble cujo print() é um “método aprimorado que os torna mais fáceis de usar com grandes conjuntos de dados contendo objetos complexos”.
Veja o seguinte exemplo:
Primeiramente, imagine que você queira calcular a média de mpg por cyl dos dados mtcars (nativos do R), bastaria utilizar a sequência de códigos:
mtcars %>% # input data
group_by(cyl) %>% # split
summarise(media = mean(mpg)) # apply/combine## # A tibble: 3 x 2
## cyl media
## <dbl> <dbl>
## 1 4 26.7
## 2 6 19.7
## 3 8 15.1Vejamos a seguir o uso da estratégia em situações mais complexas
Em ajustes de modelos
E se precisássemos calcular algo mais elaborado, como por exemplo ajustar \(k=3\) regressões lineares: \(y_k= b_{0_k} + b_{1_k}*x_k\) (com \(y_k=\) mpg, \(x_k=\)disp para cada \(k=\)cyl) para estudar os coeficientes estimados, o que aconteceria se utilizássemos o código abaixo ?
Spoiler: Note que pelo fato da saída da função lm não retornar apenas uma única variável para sumarizar obteremos um Error:
mtcars %>% # input data
group_by(cyl) %>% # split
summarise(lm = lm(mpg ~ disp)) # apply/combine## Error: Problem with `summarise()` input `lm`.
## x Input `lm` must be a vector, not a `lm` object.
## ℹ Input `lm` is `lm(mpg ~ disp)`.
## ℹ The error occurred in group 1: cyl = 4.O erro nos diz: “A coluna lm deve ter o comprimento 1, não 12” ou seja, o resultado precisa ser um valor de resumo e não todo o resultado do ajuste dos modelos.
Agora vejamos utilizando a abordagem split-apply-combine que irá nos permitir aplicar qualquer tipo de função nos dados agrupados por pela variável cyl:
as_tibble(mtcars) %>% # input data
nest(-cyl) %>% # split
mutate(lm = map(data, ~lm(mpg ~ disp, data = .x) %>% broom::tidy())) %>% # apply
unnest(lm) # combine## # A tibble: 6 x 7
## cyl data term estimate std.error statistic p.value
## <dbl> <list> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 6 <tibble [7 × 10]> (Intercept) 19.1 2.91 6.55 0.00124
## 2 6 <tibble [7 × 10]> disp 0.00361 0.0156 0.232 0.826
## 3 4 <tibble [11 × 10]> (Intercept) 40.9 3.59 11.4 0.00000120
## 4 4 <tibble [11 × 10]> disp -0.135 0.0332 -4.07 0.00278
## 5 8 <tibble [14 × 10]> (Intercept) 22.0 3.35 6.59 0.0000259
## 6 8 <tibble [14 × 10]> disp -0.0196 0.00932 -2.11 0.0568Com o auxílio do pacote broom obtemos saídas de dados arrumados e juntamos os resultados finais da regressão em uma única tabela de maneira prática.
Na construção de gráficos
Veja um outro exemplo de uso aplicando uma função para criar gráficos, agora com ggplot:
library(ggplot2)
library(gridExtra)
plot_list <-
mtcars %>% # input data
nest(-cyl) %>% # split/apply ↓
mutate(plots = map(data, ~ggplot(.x, aes(x=disp, y=mpg))+geom_point()+geom_smooth(method = "lm"))) %$%
plots # magrittr
# Combine para printar:
invoke(grid.arrange,plot_list, ncol=1) # ou: grid.arrange(grobs = plot_list, ncol=1)
# Combine para salvar:
walk2(paste0("plot",1:3,".png"), plot_list, ~ggsave(.x,.y))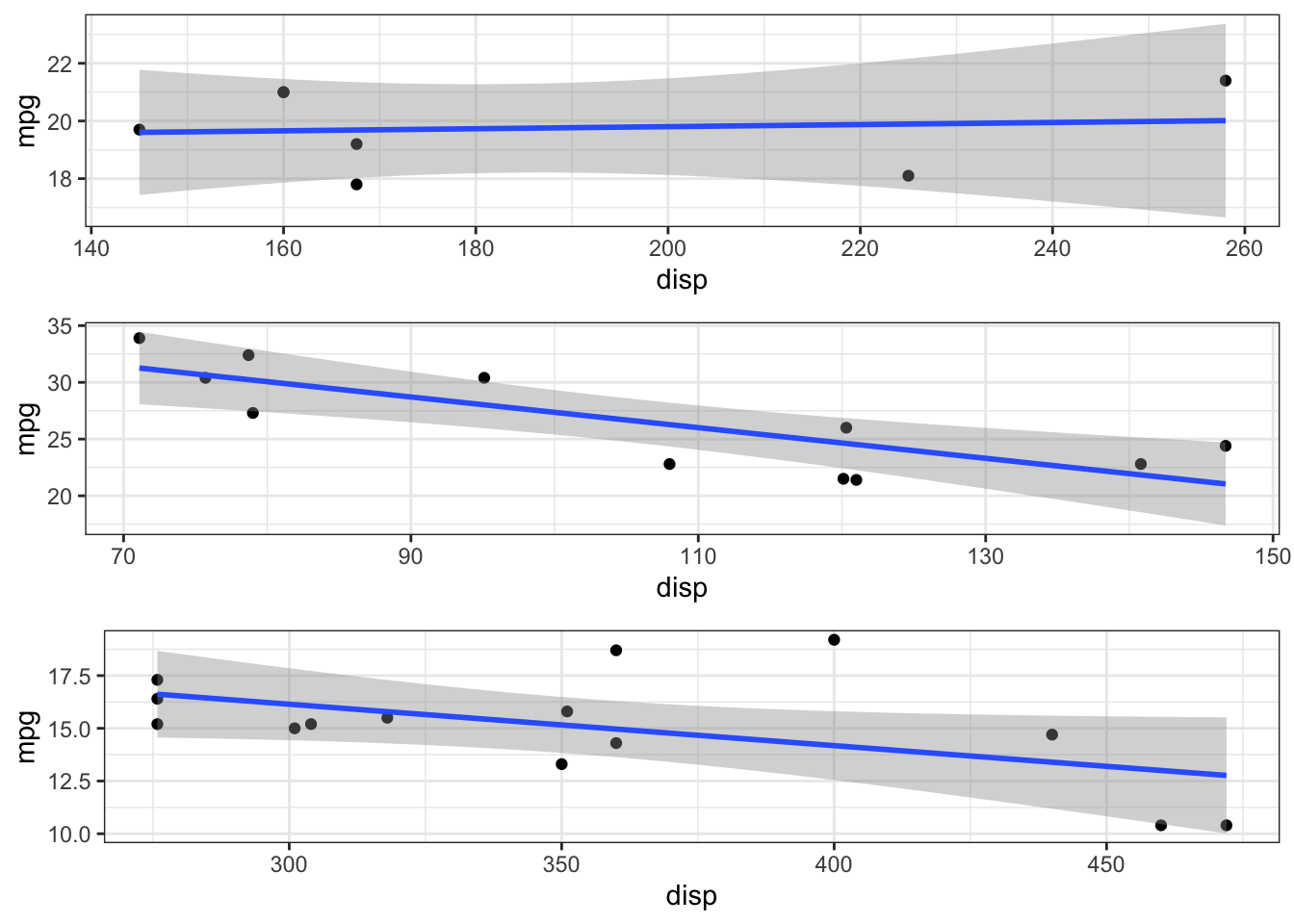
Criando tabelas
Por fim, um exemplo utilizando o pacote flextable.
Utilizaremos a função flextable_custom() que adaptei para gerar uma tabela já customizada com o pacote flextable e a função save_flextable() inspirada em uma pergunta que fiz no stackoverflow sobre Como salvar uma tabela flextable como png no R?.
Veja:
library(flextable)
source("https://raw.githubusercontent.com/gomesfellipe/functions/master/flextable_custom.R")
source("https://raw.githubusercontent.com/gomesfellipe/functions/master/save_flextable.R")
tabela_list <-
head(mtcars,7) %>% # input data
nest(-cyl) %$% data %>% # apply
map(~flextable_custom(.x)) # apply / combine
# Veja a tabela:
tabela_list[[1]]
# Combine para salvar:
walk2(paste0("tab",1:3,".png"), tabela_list, ~save_flextable(.y,.x))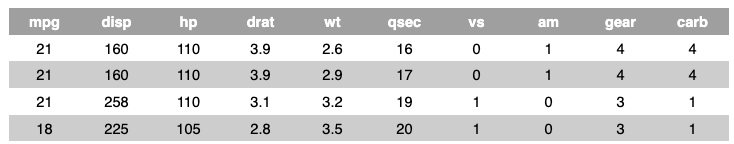
Conclusão
Vimos aqui como funciona a estratégia e alguns exemplos de uso, porém, existem infinitas outras aplicações para esse tipo de abordagem com os dados arrumados. Dependendo da tarefa esta abordagem pode ser bem produtiva e poupar muitas linhas de código!
Referências
Além das referências deixarem aqui algumas sugestões de leitura:






























Share this post
Twitter
LinkedIn
Email