Prevendo a qualidade do sono utilizando Machine Learning
Utilizaremos dados reais coletados pelo celular para gerar previsões a partir de uma pequena base de dados com target desbalanceada

Qualidade de sono? 🤨
Sim, exatamente! Neste post analisaremos dados de um tracking que venho fazendo desde 2017 com informações relacionadas à um sono de qualidade.
Boas noites de sono nos tornam mais felizes, mais saudáveis, mais inteligentes, mais dispostos e evita problemas de cansaço, falta de concentração, depressão e ansiedade.
Resumindo, a nossa qualidade de vida está diretamente ligada à qualidade do nosso sono, pois ao dormir nosso corpo realiza funções extremamente importantes como por exemplo o fortalecimento do sistema imunológico, secreção e liberação de hormônios, consolidação da memória, entre outras1.
Alguns fatores podem auxiliar a determinar se uma noite foi bem dormida como por exemplo: a regularidade do horário de dormir e de acordar, a frequência cardíaca (bpm), número de passos dados no dia, tempo na cama, tempo antes de dormir, ronco, tipo de clima etc..
Felizmente, existe um aplicativo chamado Sleep Cycle que é capaz de trackear todas essas informações durante o uso do app, dentre outras funcionalidades. Desde 2017 tenho acompanhado meu sono através dele, principalmente pela funcionalidade de rastreio dos padrões de sono para despertar durante sua fase mais leve, sem um despertador convencional e tenho curtido bastante!
A proposta principal do aplicativo é monitorar os sinais do corpo para nos despertar suavemente quando estivermos no estágio de sono mais leve possível, pois acordar durante o sono leve é como acordar naturalmente descansado!
Como funciona aplicativo Sleep Cycle? 
Tradução livre de How Sleep Cycle works:
"O funcionamento básico desse aplicativo se baseia que nos mexemos predominantemente durante o sono leve. Já durante o sono pesado, os músculos tendem a permanecer relaxados, e em sono REM a movimentação muscular abaixo do pescoço fica paralizada.
Assim sendo é possível selecionar um horário que gostaria de acordar, como de 6:30 até 7:00, e o aplicativo rastreará os movimentos na cama para acordar apenas quando entrar em sono leve durnte este período.
Dessa forma, estaríamos aumentando as chances de acordar mais bem-disposto, já que seu sono foi interrompido em uma fase mais leve de descanso."
Vejamos dois gráficos que exemplificam dois dos possíveis cenários de uma noite de sono:
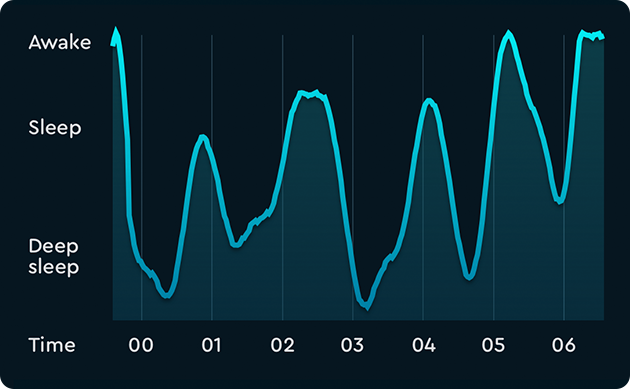 Os picos representam os ciclos do sono, incluindo todas as fases do sono.
Os picos representam os ciclos do sono, incluindo todas as fases do sono.
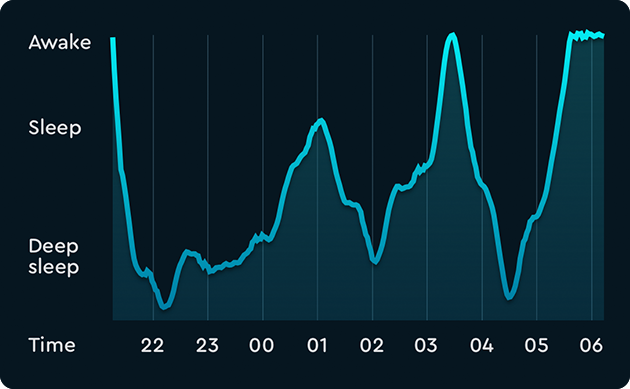 Ciclos de sono mais irregulares, onde o usuário provavelmente não dormiu tão bem como em nosso primeiro exemplo.
Ciclos de sono mais irregulares, onde o usuário provavelmente não dormiu tão bem como em nosso primeiro exemplo.
Esta é a principal informação coletada no aplicativo e que permite um “despertar tranquilo”!
Objetivo 🎯
Apesar do aplicativo captar diversos dados sobre a noite de sono, o “humor ao acordar” é uma informação fornecida pelo usuário assim que desativa o alarme, quando a seguinte tela é exibida:

- Onde:
-
- 😃 (Bom)
-
- 😑 (Ok)
-
- 😡 (Mau)
Como houveram diversos dias em que utilizei o aplicativo mas não assinalei o humor (seja por ter desativado o recurso por algum tempo ou simplesmente por ter ignorado 😅) vamos trabalhar para responder a seguinte pergunta:
Qual foi a probabilidade de ter acordado de mal humor durante o período de tracking do app, nos dias cujo esse dado é faltante?
Onde mal humor será a classe positiva da target, traduzido nos dados da seguinte forma:
\[ mood= \begin{cases} Bom, & \text{se}\ mood = Bom \\ Ruim, & \text{c.c}\ \end{cases} \]
Logo, mood será binária, avaliando se o humor foi Bom ou Ruim ao acordar, onde Ruim a combinação do status 😑 (Ok) e 😡 (Mau) e será a classe mais importante para controlar os erros de previsão.
Tomei a liberdade de fazer essa transformação pois desde o início do uso do app, marco como Ruim apenas quando realmente não descansei de forma satisfatória. Isso pode ter ocorrido por diversos fatores, como por exemplo: acordar após um pesadelo; acordar com barulho da rua ou de casa; acordar meio doente ou passando mal e por ai vai..
Por enquanto, estas informações serão suficientes. Vejamos na análise exploratória como se apresenta a variável target e quais dados disponíveis para atingir tal objetivo.
Explorar dados 🔎
Carregar as dependências:
library(tidyverse) # datascience toolkit
library(lubridate) # manipule date
library(patchwork) # grid ggplot
library(tidymodels) # machine learning toolkit
library(reactable) # print tables
library(treesnip) # lightgbm
# Definir tema para ggplot
theme_set(theme_bw()) Vamos carregar funções que foram desenvolvidas ao longo das análises para facilitar tanto na apresentação dos resultados quanto na portabilidade dos códigos (bastando pequenos ajustes para “reciclá-los” ♻️):
(Clique aqui para exibir as funções customizadas)
# Para o print de tabelas
print_table <- function(x, round=0, evalue_model = F, ...){
if(round>0) x <- x %>% mutate_if(is.numeric, ~round(.x, round))
if(evalue_model == T){
reactable::reactable(x, striped = T, bordered = T,
highlight = T, pagination = F,
width = 800,
defaultColDef = colDef(minWidth = 85),
defaultSorted = list(auc_pr = "desc"),
columns = list(
model = colDef(minWidth = 110),
tp = colDef(minWidth = 40),
fp = colDef(minWidth = 40),
fn = colDef(minWidth = 40),
tn = colDef(minWidth = 40)),
...)
}else{
reactable::reactable(x, striped = T, bordered = T, width = 800,
highlight = T, pagination = F, ...)
}
}
# Graficos de features numericas
plot_num <- function(data, num_feature,
title = NULL, bins = 30, legend = NULL){
if(is.null(title)) title = num_feature
data = data %>% filter(!is.na(mood))
p_shapiro = round(shapiro.test(data$air_pressure_pa)$p.value, 5)
p1 <-
data %>%
ggplot(aes_string(x = num_feature, fill = "mood"))+
geom_histogram(aes(y=..density..), bins = bins, alpha = 0.5,
show.legend = ifelse(!is.null(legend), T, F))+
geom_density(alpha = 0.5,
show.legend = ifelse(!is.null(legend), T, F))+
labs(y = "", x= "", title = title)+
scale_fill_viridis_d(end = 0.8, direction = 1)
if(!is.null(legend)){
p1 = p1 + theme(legend.position = legend)
}
p2 <-
data %>%
ggplot(aes_string(x = num_feature))+
geom_boxplot(aes(y = "", color = mood),
show.legend = F)+
labs(y = "", x= "",
caption = paste0("Shapiro-Wilk normality test: ",
ifelse(p_shapiro == 0, "P<0.05", p_shapiro) ))+
scale_color_viridis_d(end = 0.8, direction = 1)
p1 / p2 + plot_layout(heights = c(4/5, 1/5))
}
# Graficos de features categoricas
plot_cat <- function(data, cat_feature, title = NULL, label = TRUE, legend = NULL){
data = data %>% filter(!is.na(mood))
valor_p = round(chisq.test(data %>% pull(cat_feature),
data$mood,
simulate.p.value = T)$p.value, 5)
to_plot = data %>%
count(!!as.name(cat_feature), mood) %>%
group_by(!!as.name(cat_feature))
final_plot = to_plot %>%
mutate(prop = n/sum(n),
lab = paste0(round(prop*100, 2), "%")) %>%
ggplot()+
geom_bar(aes_string(x = cat_feature, y = "n", fill = "mood"),
stat = "identity", alpha = 0.7,
position = position_dodge2(0.9),
show.legend = ifelse(!is.null(legend), T, F))+
scale_fill_viridis_d(end = 0.8, direction = 1)+
labs(title = title, y = "",
caption = paste0("Pearson's Chi-squared test: ", valor_p))
if(label == TRUE){
final_plot = final_plot+
geom_label(aes_string(x = cat_feature, y = "n", label = "lab"),
position = position_dodge2(0.9), show.legend = F)
}
if(!is.null(legend)){
final_plot = final_plot + theme(legend.position = legend)
}
return(final_plot)
}
# Grafico interativo de features temporais
plot_dygraph <- function(x, order.by, feature, title = NULL){
x %>%
xts::xts(order.by = order.by) %>%
.[,feature] %>%
dygraphs::dygraph(main = title) %>%
dygraphs::dyRangeSelector()
}
# Calcula o ponto de corte que maximiza a funcao f beta
threshold_max <- function(x){
fbeta <- function(precision, recall){
(beta+1)*(precision*recall)/(beta*(precision+recall))
}
# https://machinelearningmastery.com/fbeta-measure-for-machine-learning/
# F05: + precision - recall
# F1 : + precision + recall
# F2 : - precision + recall
beta = 0.5
x %>%
pr_curve(mood, .pred_Ruim) %>%
mutate(fbeta = fbeta(precision, recall) ) %>%
filter(fbeta == max(fbeta, na.rm = T))
}
# Plot da matriz de confusao e da funcao das funcoes de densidade estimadas
conf_mat_plot <- function(x, null_model = FALSE){
trs <- threshold_max(x)$.threshold
if(null_model==FALSE){
x <- x %>%
mutate(.pred_class = ifelse(.pred_Ruim >= trs, "Ruim", "Bom") %>%
factor(levels = c("Ruim", "Bom"), ordered = TRUE))
}
p1 <-
x %>%
select(.pred_class, mood) %>%
table() %>%
conf_mat() %>%
autoplot(type = "heatmap")+
labs(title = "Matriz de Confusao",
subtitle = paste0("Threshold max F0.5: ", round(trs, 4)))
p2 <-
x %>%
ggplot() +
geom_density(aes(x = .pred_Ruim, fill = mood),
alpha = 0.5)+
labs(title = "Distribuições de probabilidade previstas",
subtitle = "por classe")+
scale_x_continuous(limits = 0:1)+
geom_vline(aes(xintercept = trs, color = "threshold max F0.5"), linetype = 2) +
scale_color_manual(name = "", values = c(`threshold max F0.5` = "red"))+
scale_fill_viridis_d(end = 0.7, direction = 1)
p1 | p2
}
# Conjunto de metricas utilizadas para avaliar os modelos
evalue_model <- function(x, model = "", null_model=FALSE){
trs <- threshold_max(x)$.threshold
if(null_model==FALSE){
x <- x %>%
mutate(.pred_class = ifelse(.pred_Ruim >= trs, "Ruim", "Bom") %>%
factor(levels = c("Ruim", "Bom"), ordered = TRUE))
}
cm <- x %>%
select(.pred_class, mood) %>%
table()
tibble(
model = model,
tp = cm[1,1],
fp = cm[1,2],
fn = cm[2,1],
tn = cm[2,2],
auc_roc = yardstick::roc_auc(x, mood, `.pred_Ruim`)$.estimate,
auc_pr = yardstick::pr_auc(x, mood, `.pred_Ruim`)$.estimate,
logloss = yardstick::mn_log_loss_vec(x$mood, x$.pred_Ruim),
f1 = yardstick::f_meas_vec(x$mood, x$.pred_class, beta = 1),
f05 = yardstick::f_meas_vec(x$mood, x$.pred_class, beta = 0.5),
f2 = yardstick::f_meas_vec(x$mood, x$.pred_class, beta = 2),
precision = yardstick::precision_vec(x$mood, x$.pred_class),
recall = yardstick::recall_vec(x$mood, x$.pred_class),
trs_fbeta = trs
)
}
plot_auc <- function(x){
p1 <-
x %>%
group_by(model) %>%
roc_curve(mood, .pred_Ruim) %>%
ggplot(aes(x = 1 - specificity, y = sensitivity, color = model)) +
geom_line(size = 1, alpha = 0.5, show.legend = F) +
geom_abline(lty = 2, alpha = 0.5, color = "gray50", size = 1.3)+
labs(title = "AUC")+
scale_color_viridis_d(direction = 1)
p2 <-
x %>%
group_by(model) %>%
pr_curve(mood, .pred_Ruim) %>%
ggplot(aes(x = recall, y = precision, color = model)) +
geom_line(size = 1.15, alpha = 0.5) +
# geom_abline(slope = -1, intercept = 1, lty = 2, alpha = 0.5, color = "gray50", size = 1.2)+
labs(title = "PR AUC")+
theme(legend.position = "right")+
scale_color_viridis_d(direction = 1)
(p1 | p2)
}
Importar os dados obtidos no app SleepCycle e padronizar nomes das colunas:
sleep <- read_csv2("sleepdata.csv") %>% janitor::clean_names(case = "snake")A seguir, uma tabela com uma descrição do conteúdo de cada coluna:
(Código do objeto dic)
dic <-
tibble(
`Coluna` = sleep %>% colnames(),
`Descrição curta` =
c("Inicio",
"Fim",
"Qualidade do Sono",
"Regularidade",
"Humor",
"Frequência cardíaca (bpm)",
"Passos",
"Modo de alarme",
"Pressão do Ar (Pa)",
"Cidade",
"Movimentos por hora",
"Tempo na cama (segundos)",
"Tempo adormecido (segundos)",
"Tempo antes de dormir (segundos)",
"Início da janela",
"Fim da janela",
"Ronco",
"Hora do ronco",
"Temperatura (°C)",
"Tipo de clima",
"Notas"),
`Descrição detalhada` =
c("Início do monitoramento",
"Fim do monitoramento",
"Qualidade do sono é baseada em: tempo que passa a dormir, movimentos durante a noite e momentos em que está totalmente desperto",
"Informa sobre a regularidade do horário de dormir e de acordar durante um período de tempo. Quanto maior, mais regular tem sido o horário de acordar e dormir e isso pode resultar em um sono melhor",
"Humor informado no app ao acordar:
\U0001F603 (Bom), \U0001F611 (Ok), \U0001F621 (Mau), \U00026D4 (Não informado)",
"-",
"Quantos passos dá por dia (bom a partir de 10.000 passos por dia)",
"Alarme ligado ou apenas monitoramento",
"-",
"-",
"-",
"-",
"-",
"-",
"Início do modo soneca",
"Fim do modo soneca",
"Detector de ruídos (pode captar outros barulhos que não seja ronco)",
"-",
"-",
"-",
"Alguma nota ao acordar"
),
Target = c(rep("",4), "🎯", rep("", 16))
)dic %>% print_table(columns = list(Target = colDef(minWidth = 35)))
Limpeza e preparação dos dados
Vamos realizar uma limpeza inicial, preparando os dados para possibilitar as análise em um objeto tibble minimamente arrumado:
sleep <- sleep %>%
# fix target
mutate(mood = case_when(mood == "Bom" ~ "Bom",
mood == "Mau" ~ "Ruim",
mood == "Ok" ~ "Ruim",
is.na(mood) ~ NA_character_),
mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
# fix window
mutate_at(c("window_start", "window_stop"),
~ifelse(is.na(.x), end, .x)) %>%
# fix string %
mutate_at(c("sleep_quality", "regularity"),
~ .x %>% str_remove("%") %>% as.numeric() ) %>%
# fix heart_rate_bpm e criar bug indicator
mutate(heart_rate_bug = ifelse(heart_rate_bpm == 0, "sim", "nao")) %>%
mutate(heart_rate_bpm = ifelse(heart_rate_bpm == 0,
NA_integer_, heart_rate_bpm)) %>%
# fix dados de soneca
mutate(snore_time = as.numeric(snore_time),
did_snore = ifelse(did_snore == TRUE, "sim", "nao")) %>%
# fix para numerico
mutate_at(c("time_before_sleep_seconds",
"time_asleep_seconds",
"time_in_bed_seconds"),
~as.numeric(.x) ) %>%
# fix movements_per_hour para double
mutate(movements_per_hour = as.double(movements_per_hour)) %>%
# fix weather_type
mutate(weather_type =
factor(weather_type,
levels = c("No weather", "Rain", "Rainy showers", "Cloudy",
"Partly cloudy", "Fair", "Sunny"),
ordered = TRUE)) %>%
mutate_at(c("weather_temperature_c", "air_pressure_pa"),
~ as.numeric(.x) %>% if_else(. == 0, NA_real_, .)) %>%
# remover unused columns
select(-one_of(c("city", "notes"))) %>%
select(mood, everything()) %>%
arrange(end)Qual a estrutura geral dos dados? Será que existe algum padrão nos dados ausentes?
sleep %>%
arrange(end) %>%
mutate(Date = as.Date(end))%>%
# complete(Date = seq.Date(min(Date), max(Date), by="day")) %>%
visdat::vis_dat() 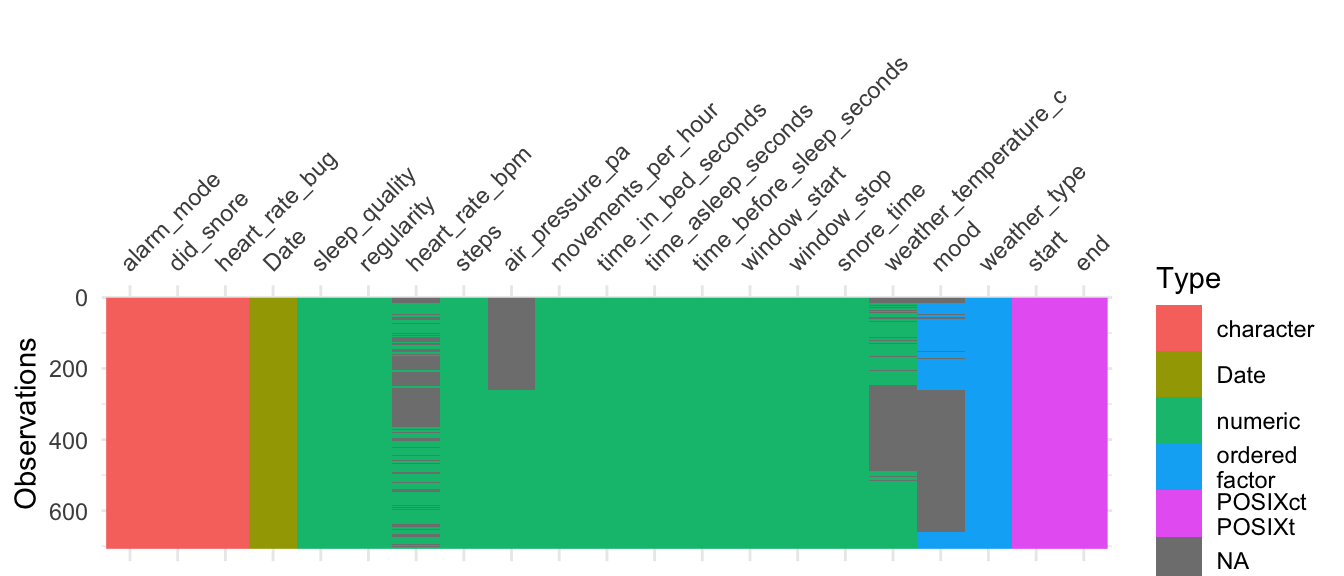
Os dados ausentes ocorrem tanto espalhados (heart_rate_bpm) quanto em sequência (air_pressure_pa, weather_temperature_c, mood) portando, adotaremos as seguintes estratégias para inputar dados ausentes:
air_pressure_pa: Será obtidos no site data.rio/datasets e caso ainda exista dados ausentes, será preenchido com as médias móveis dos últimos 7 dias;weather_temperature_c: Mesma estratégia do item (1);heart_rate_bpm: Cálculo das médias móveis dos últimos 7 dias;mood: Como é a target, as instâncias aondeis.na(mood)serão retidas para estimação após o ajuste do modelo.
Imputar dados de fontes externas
O preenchimento das features air_pressure_pa, weather_temperature_c serão realizados a partir do download de dados públicos do Rio de Janeiro no link: data.rio/datasets. Para obter este dado utilizaremos a função get_rj_data() desenvolvida para este post, que está omitida mas para quem tiver interesse basta conferir clicando no item abaixo:
(Código da função get_rj_data())
get_rj_data <- function(){
if(!file.exists("rj_data.rds")){
url <- "https://opendata.arcgis.com/datasets/5b1bf5c3e5114564bbf9b7a372b85e17_2.csv?outSR=%7B%22latestWkid%22%3A4326%2C%22wkid%22%3A4326%7D"
download.file(url, "rj_data.csv")
rj_data <- readr::read_csv("rj_data.csv")
saveRDS(rj_data, "rj_data.rds")
}else{
rj_data <- readRDS("rj_data.rds")
}
# preparar dados de pressao atmosferica e temperatura no periodo desejado
rj_data <- rj_data %>%
mutate(Data = ymd_hms(Data)) %>%
filter(Data >= min(sleep$start) & Data <= max(sleep$start)) %>%
group_by(Data = as.Date(Data)) %>%
summarise(air_pressure_pa = mean(Pres/10, rm.na=T),
weather_temperature_c = mean(Temp, rm.na=T))
return(rj_data)
}Com acesso aos dados, hora de combinar as bases e preencher os dados faltantes:
sleep <- sleep %>%
mutate(Data = as.Date(start)) %>%
# to numeric
mutate_at(c("weather_temperature_c", "air_pressure_pa"),
~ as.numeric(.x) %>% if_else(. == 0, NA_real_, .)) %>%
# join Rio data
left_join(get_rj_data() , by = c("Data")) %>%
# fill with new data
mutate(air_pressure_pa = ifelse(is.na(air_pressure_pa.x),
air_pressure_pa.y,
air_pressure_pa.x)) %>%
mutate(weather_temperature_c = ifelse(is.na(weather_temperature_c.x),
weather_temperature_c.y,
weather_temperature_c.x)) %>%
# remove aux columns
select(-air_pressure_pa.x, -air_pressure_pa.y,
-weather_temperature_c.x, -weather_temperature_c.y,
-Data)Insights
Nesta seção vamos responder algumas perguntas com dados!
start e end
Qual a média mensal de horas dormidas e que horas costumo acordar, em média, mensalmente ao longo desses anos?
(Código para gráfco abaixo)
dy1 <- sleep %>%
complete(start = seq.Date(min(as.Date(start)), max(as.Date(start)), by="day")) %>%
mutate(dif_sleep_hours = as.numeric(end - start)/60) %>%
mutate(dif_sleep_hours = zoo::rollmean(dif_sleep_hours, k = 30, fill = NA)) %>%
plot_dygraph(order.by = .$start, feature = 'dif_sleep_hours')
dy2 <- sleep %>%
complete(start = seq.Date(min(as.Date(start)), max(as.Date(start)), by="day")) %>%
mutate(end_hour = hour(end)) %>%
mutate(end_hour = zoo::rollmean(end_hour, k = 30, fill = NA)) %>%
plot_dygraph(order.by = .$start, feature = 'end_hour')
Tempo dormindo (em horas) Média móvel 30 dias
O tempo que passa dormindo parece variar (em média) em torno de 6 à 7 horasHora que acorda Média móvel 30 dias
O pico no início no gráfico corresponde ao penúltimo semestre da facultado. No final de 2017 comecei a trabalhare passei a acordar mais cedo⚠️ Note que existem alguns espaços vazios, que correspondem aos dias que o app não foi utilizado.
window_start e window_stop
Quanto tempo costumo usar o modo “soneca” ao longo da semana? E aos finais de semana?
(Código para gráfco abaixo)
p <- sleep %>%
mutate(mood = ifelse(is.na(mood), "NA", as.character(mood)) %>%
factor(levels = c("Ruim", "NA", "Bom"))) %>%
mutate(nap_minutes = (window_stop - window_start) / 30,
final_de_semana = lubridate::wday(start) %in% c(1, 7)) %>%
count(mood, final_de_semana, nap_minutes) %>%
group_by(mood, final_de_semana) %>%
mutate(
fnap_minutes = case_when(
nap_minutes == 0 ~ "Sem modo soneca",
nap_minutes == 20 ~ "20 minutos",
nap_minutes == 30 ~ "30 minutos",
nap_minutes == 60 ~ "1 hora"),
fnap_minutes = reorder(fnap_minutes, nap_minutes),
final_de_semana = ifelse(final_de_semana == T, "Final de semena", "Dia de semana"),
label = paste0( n, " (", round(n/sum(n)*100, 2), "%)")
) %>%
ggplot(aes(x = fnap_minutes, y = n, label = label, fill = mood))+
geom_bar(stat = "identity", alpha = 0.8)+
scale_fill_viridis_d(end = 0.7, direction = 1)+
# ggrepel::geom_label_repel(aes(label = label))+
labs(x = "", y = "")+
facet_wrap(~final_de_semana)+
theme(axis.text.x = element_text(angle = 30, hjust=1))
p %>% plotly::ggplotly() %>% plotly::config(displayModeBar = F)Como era de se esperar, os dias em que mood=="Ruim" ocorrem mais quando o modo soneca não é ativado pois acaba mesmo sendo menos propício a voltar a dormir. Outro detalhe é que muitas vezes usei o soneca por um tempo muito prolongado! (😱 pelo menos mood=="Bom" na maioria desses casos!)
Já nos finais de semana, ocorre pouquíssimo mood== "Ruim" e praticamente não há uso do alarme e quando há, não utiliza soneca.
weather_type e alarm_mode
Será que o humor ao acordar esta relacionado com o tipo de clima ou com o modo utilizado no alarme?
p1 <- plot_cat(sleep, cat_feature="weather_type",
title = "Tipo de clima", label = F)+
theme(axis.text.x = element_text(angle = 30, hjust=1))
p2 <- plot_cat(sleep, cat_feature="alarm_mode",
title = "Modo de alarme", label = F, legend = "right")
p1 | p2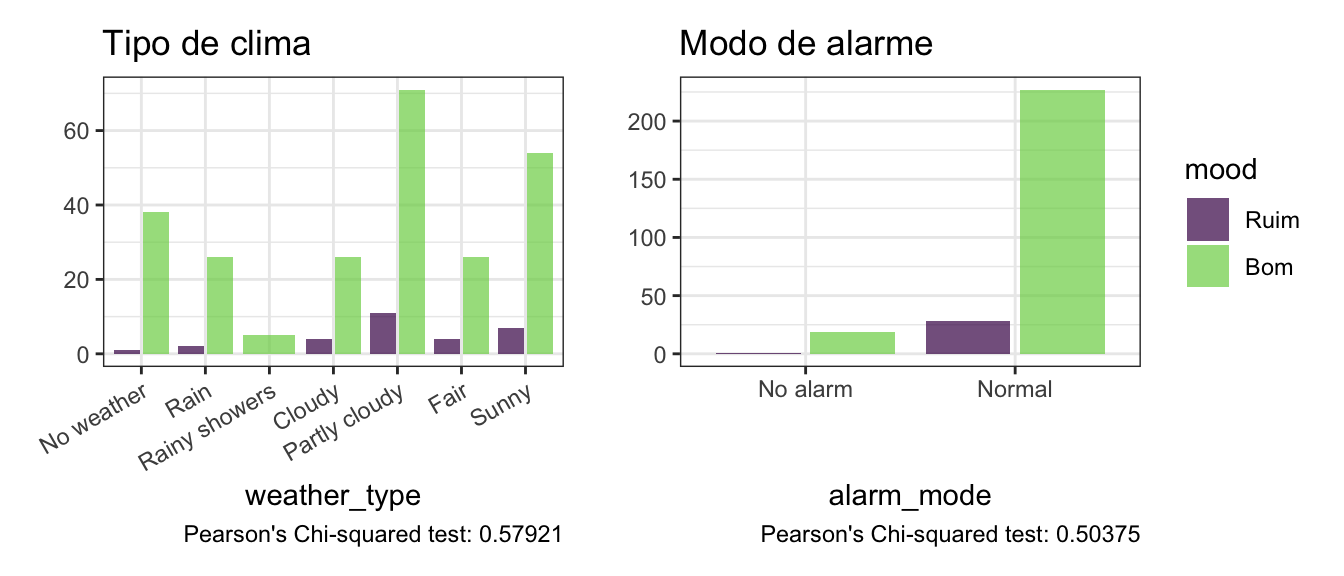
Nota-se que não existem evidências estatisticas para afimar que essas features (sozinhas) estão associadas à target, porém como serão utilizados modelos baseados em árvores que experimentam diversas combinações de features, vamos manter na base e deixar o modelo decidir como usar.
sleep_quality e time_in_bed_seconds
A qualidade de sono e o tempo da cama estão normalmente distribuídos em torno de uma média?
p1 <- sleep %>% plot_num("sleep_quality")
p2 <- sleep %>% plot_num("time_in_bed_seconds", legend = "right")
p1 | p2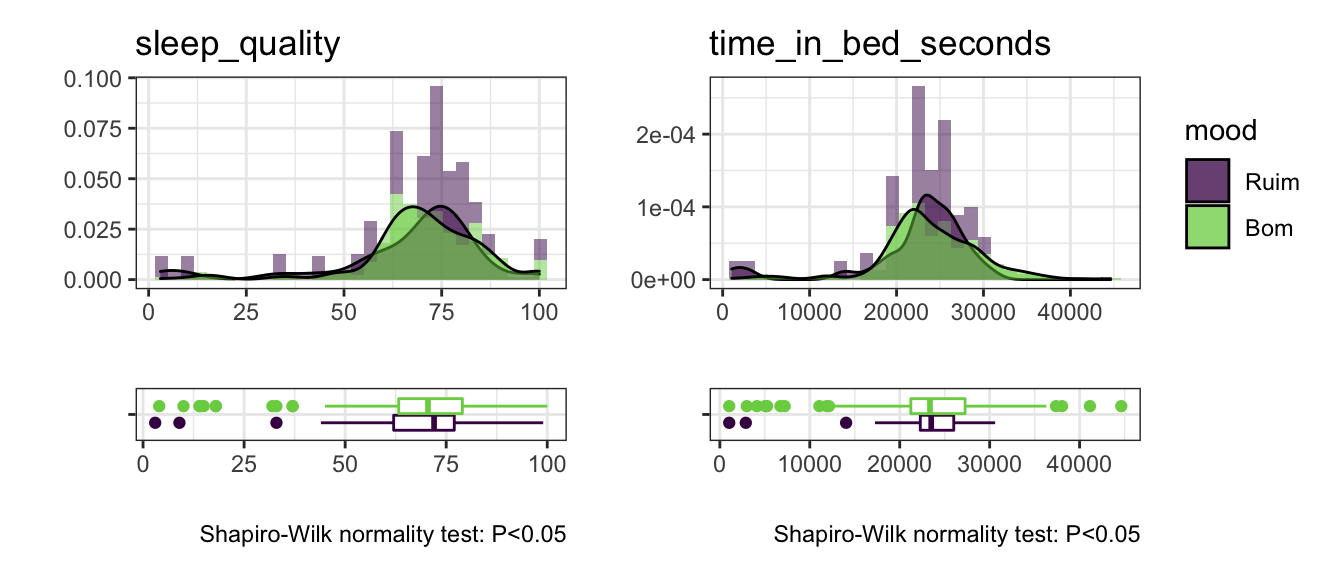
Existem alguns registros em que o tempo na cama é menor que 10.000 segundos (~3horas) o que corresponde aos pequenos cochilos que registrei no app. Não foram muitos registros mas talvez seja útil na modelagem pois existem ocorrências de humor (mood) Bom e Ruim ali.
Como a correlação de spearman entre estas duas feautures é muito alta (0.8705) é possível notar que baixa qualidade do sono esta altamente correlacionada com o tempo na cama.
Mais uma pergunta sobre estas features: Como a média mensal da qualidade do sono e do tempo na cama em horas estão distribuídos ao longo do tempo?
(Código para gráfco abaixo)
dy1 <- sleep %>%
complete(start = seq.Date(min(as.Date(start)),
max(as.Date(start)), by="day")) %>%
mutate(sleep_quality = zoo::rollmean(sleep_quality, k = 30, fill = NA)) %>%
plot_dygraph(order.by = .$start, feature = 'sleep_quality')
dy2 <- sleep %>%
complete(start = seq.Date(min(as.Date(start)),
max(as.Date(start)), by="day")) %>%
mutate(time_in_bed_seconds =
zoo::rollmean(time_in_bed_seconds, k = 30, fill = NA)) %>%
mutate(time_in_bed_seconds = time_in_bed_seconds / 60 / 60) %>%
plot_dygraph(order.by = .$start, feature = 'time_in_bed_seconds')
Qualidade do sono Média móvel 30 dias
Parece que a qualidade do sono vem aumentando desde final de 2019, mantendo um patamar semlhante ao final e 2018.Tempo na cama em horas Média móvel 30 dias
O tempo na cama varia entre 6 à 7 horas (Apesar de alguns picos em 2020, provavelmente por conta da pandemia do corona virus quando estabeleceu-se o home office)Reter dados
Antes de iniciar o processo de modelagem, será necessário reter dados aonde mood é NA, pois faremos as previsões nestes dados apenas após o ajuste e seleção do modelo final.
new_sleep <- sleep %>% filter(is.na(mood))
sleep <- sleep %>% filter(!is.na(mood))Modelagem 🚀
Hora de criar alguns modelos para estimar a probabilidade das classes da target: mood.
Neste caso, as questões são as seguintes:
- Qual a classe mais importante?
- Qual a métrica será utilizada para selecionar os modelos?
- Qual será o threshold?
- Qual será estratégia para lidar com o desbalanceamento?
- Quais métricas serão monitoradas?

A classe mais importante para nossa previsão é a positiva, ou seja, mood=="Ruim". Sendo assim desejamos evitar falsos positivos.
A métrica utilizada para selecionar os modelos será a logloss. Esta é uma métrica probabilistica que foca na incerteza que o modelo tem nas previsões e penaliza as previsões que estão erradas2.
Após calibrar a probabilidade, estabeleceremos um ponto de corte que maximizar a medida F-Beta, (que é uma abstração da medida F1, média harmônica entre Precision e Recall) onde Beta = 0.5. Essa medida tem o efeito de aumentar a importância da Precision e diminui a importância do Recall. 3
Parra lidar com o desbalanceamento da target, utilizaremos o método de undersampling chamado ** Tomek Links **4. Este método faz uma amostragem da classe majoritária de forma “mais esperta” que uma simples amostragem aleatória.
Por fim, a principal métrica que será monitorada será a AUC-PR5 (Area Under Precision Recall Curve). Ela é uma espécie de AUC que cálculada a área sobre a Precision x Recall. Essa métrica é preferível neste caso pois foca maisn na classe positiva e a ROC AUC tente a superestimar os valores nesse caso.
Amostragem
Para preparar os dados para modelagem vamos dividir os dados em treino (70%) e teste (30%).
set.seed(123456789)
# treino e teste
sleep_split <- initial_split(data = sleep, strata = mood, prop = 0.7)
sleep_train <- training(sleep_split)
sleep_test <- testing(sleep_split)Além disso, vamos dividir o conjunto de treino em 4 folds para obter resultados de validação cruzada. Este valor corresponde metade da quantidade em que mood=="Ruim" nos dados teste.
set.seed(123456789)
k_fold <- sleep_test %>% count(mood) %>% filter(mood=="Ruim") %>% pull(n)
sleep_folds <- sleep_train %>%
rsample::vfold_cv(v = round(k_fold/2), repeats = 10, strata = mood)A decisão de utilizar o valor de k como metade do tamanho da classe minoritária foi uma decisão pessoal, não sei se é ótima mas foi conveniente neste caso.
Como ficou dividido:
(Código para tabela abaixo)
tab <-
full_join(sleep_train %>% count(mood) %>% mutate(prop = n/sum(n)*100),
sleep_test %>% count(mood) %>% mutate(prop = n/sum(n)*100),
by = "mood") %>%
print_table(round = 2,
columns = list(
n.x = colDef(name = "N"),
prop.x = colDef(name = "(%)", align = "left"),
n.y = colDef(name = "N"),
prop.y = colDef(name = "(%)", align = "left")
),
columnGroups = list(
colGroup(name = "Train", columns = c("n.x", "prop.x")),
colGroup(name = "Test", columns = c("n.y", "prop.y"))
))
tab☠️ A quantidade reduzida de dados para teste reflete a baixa quantidade de dados no geral!
Engenharia de recursos
Hora de criar o objeto que vai conter todos os passos do pré-processamento necessário! Esse passo é muito importante pois algumas estatísticas precisam ser calculadas nos dados de treino isoladamente para não “dar pistas” para modelo sobre as informações contidas nos dados de teste, comprometendo o desempenho do modelo em novos dados.
De forma semelhante (mas não igual) ao sklearn.pipeline.Pipeline, disponível para Python, na linguagem R existe o pacote recipes que permite a criação de “receitas” com a função recipe() e que pode ser utilizada em um workflow() para treinar o modelo na sequência.
Sendo assim, algumas das operações realizadas no préprocessamento do modelo:
- Criar feature:
ano; - Criar feature:
mes; - Criar feature:
dia da semana; - Criar feature:
dia do mes; - Criar feature:
hora que acordou; - Criar feature:
final de semana; - Criar feature:
tempo dormindo;; - Criar feature:
tempo de soneca - Criar feature:
quarentena; - Inputar média movel semanal para preencher as features de
weather_temperature_ceair_pressure_pano RJ; - Transformar categóricas em dummy;
- Remover colunas com dados inválidos para modelo (timestamp);
- Preencher os dados faltantes de
heart_rate_bpmutilizando o algorítmoknncom 2 vizinhos mais próximos; - Aplicar o algoritmo Tomek Links, que é um método de undersampling.
Caso queira saber mais sobre métodos de undersampling para tratar dados desbalanceados sugiro a leitura deste excelente post! (Os códigos estão em Python porém a explicação da teoria é o que importa neste caso)
Preparar objeto recipe que contém um conjunto de etapas para pré-processamento de dados:
sleep_recipe <-
recipe(mood~., data = sleep_train) %>%
step_ordinalscore(weather_type) %>%
step_mutate(
ano = factor(year(end)),
mes = month(end),
dia_semana = wday(end) %>% ifelse(. == 7, 0, .),
dia_mes = mday(end),
end_hour = hour(end),
final_de_semana =
ifelse(lubridate::wday(start) %in% c(1, 7), "sim", "nao") %>% as.factor(),
dif_sleep_hours = as.numeric(end - start)/60,
dif_nap = as.numeric(window_stop - window_start) / 60,
quarentena = ifelse(start > dmy("20/03/2020"), "sim", "nao") %>% as.factor(),
nap_minutes = (window_stop - window_start) / 30
) %>%
step_mutate_at(c("weather_temperature_c", "air_pressure_pa"),
fn = ~ imputeTS::na_ma(.x, k = 7, weighting = "simple")) %>%
step_dummy(all_nominal(), -all_outcomes()) %>%
step_mutate_at(starts_with("ano"), # Fix 2018 nos novos dados
fn = ~ ifelse(is.na(.x), 0, .x)) %>%
step_rm(start, end, window_start, window_stop)%>%
step_knnimpute(heart_rate_bpm, neighbors = 2) %>%
themis::step_tomek(mood) %>%
prep()
# bake(sleep_recipe, new_data = NULL)Finalmente! 🥵
Com os dados devidamente preparados, vamos ligar as turbinas e partir para modelagem!
doParallel::registerDoParallel(4)Modelo Nulo (Baseline)
Este não é o tipo de modelo que serve para resolver problemas reais mas pode servir como um bom baseline (“pior que isso não fica”) pois ele vai prever apenas a classe majoritária, e com base nisso, poderemos comparar as métricas de performance do ajuste para saber se nossos modelos estão (no mínimo) performando melhor que um modelo que classifica unicamente 1 classe,
null_model <- null_model(mode = "classification") %>%
set_engine("parsnip")Definir o objeto workflow:
null_wflow_bas <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(null_model) Realizar ajuste final nos dados de treino:
null_final_fit_bas <- null_wflow_bas %>% last_fit(sleep_split) Coletar previsões nos dados de teste:
null_test_preds_bas <- collect_predictions(null_final_fit_bas)Avaliar desempenho do modelo nos dados de teste:
null_test_preds_bas %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot(null_model = T)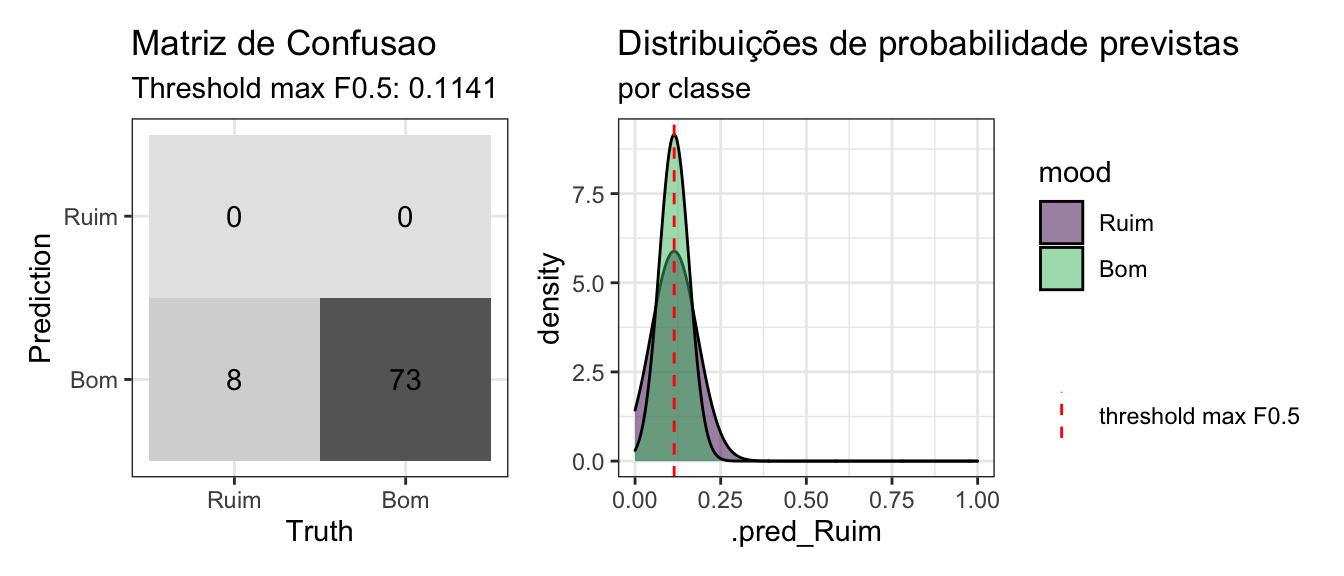
Modelo nulo pronto! Vamos para a modelagem propriamente dita!
Árvore de decisões
Este algorítimo é um ótimo ponto de partida pois possui alta explicabilidade, gerando um plot intuitivo e muito fácil de interpretar. As features que aparecem no topo são as mais importantes e cada nó seguinte é gerado a partir de regras que otimizam a divisão dos dados daquele ramo.
Existem recursos interessantes ao trabalhar com árvores, como determinar uma regra de parada ou ainda deixar a árvore crescer e depois realizar a poda. Primeiramente vamos ajusta uma árvore de decisões default e em seguida realizar algum tipo de tunning para tentar obter resultados melhores.
Default
Os parâmetros default foram definidos baseados na documentação oficial do pacote rpart em https://cran.r-project.org/web/packages/rpart/rpart.pdf e o de/para para definição dos parâmetros na página do pacote parsnip em https://parsnip.tidymodels.org/reference/decision_tree.html
tree_model_bas <- decision_tree(
cost_complexity = 0.01, # cp
tree_depth = 30, # maxdepth
min_n = 20 # minsplit
) %>%
set_engine("rpart") %>%
set_mode("classification")Definir o objeto workflow:
tree_wflow_bas <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(tree_model_bas) Ajustar modelo via validação cruzada:
tree_res_bas <- fit_resamples(
tree_wflow_bas,
sleep_folds,
metrics = metric_set(pr_auc, roc_auc, mn_log_loss),
control = control_resamples(save_pred = TRUE)
)
# Salvar "cache" da otimizacao
saveRDS(tree_res_bas, "tree_res_bas.rds")Finalizar o modelo:
# Finalizar workflow com parametros selecionados (default nesse caso)
tree_final_wflow_bas <-
finalize_workflow(
tree_wflow_bas,
select_best(tree_res_bas, metric = 'mn_log_loss')
)
# Realizar ajuste final nos dados de treino
tree_final_fit_bas <- tree_final_wflow_bas %>% last_fit(sleep_split)
# Coletar previsões nos dados de teste
tree_test_preds_bas <- collect_predictions(tree_final_fit_bas)Vejamos como ficou o modelo baseline:
(Código do objeto tre_model_bas)
tre_model_bas <-
tree_final_fit_bas$.workflow[[1]] %>%
pull_workflow_fit()
rattle::fancyRpartPlot(tre_model_bas$fit, sub = NULL, cex = 0.6)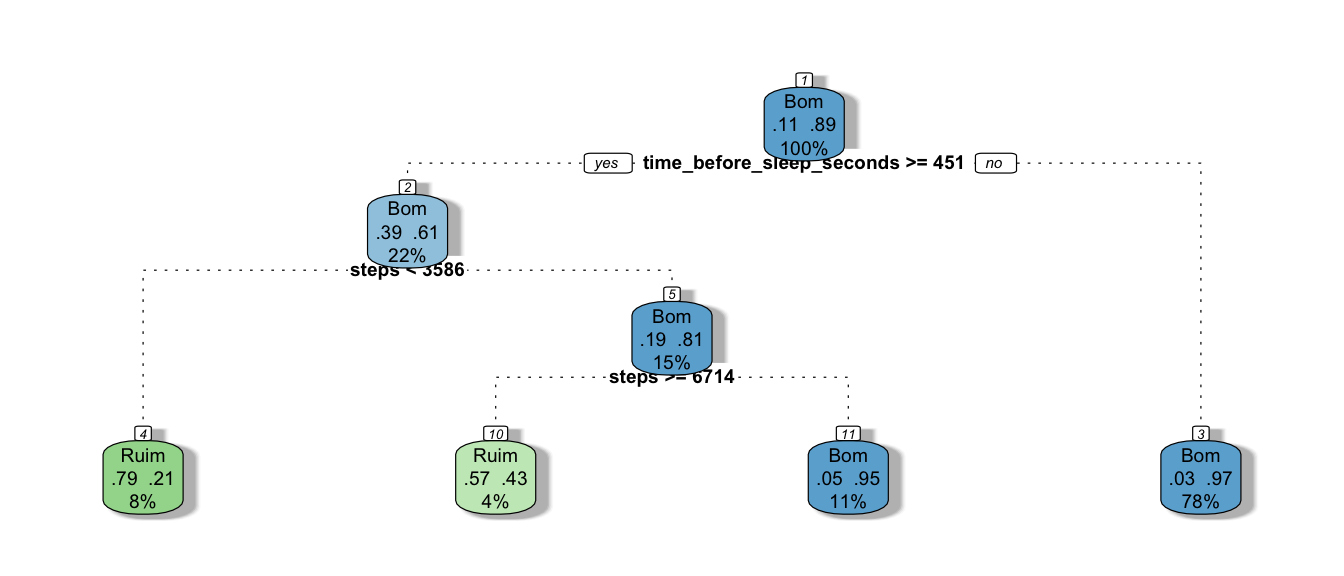
Note que o modelo default se baseia nas features time_before_sleep_seconds e steps. Talvez, com outra combinação de parâmetros seja possível conseguir um modelo uma árvore um pouco maior com resultado igual/melhor.
Como são apenas duas features, é possível visualizar os regras de classificação a partir de um gráfio de dispersão
sleep_train %>%
ggplot(aes(time_before_sleep_seconds, steps)) +
parttree::geom_parttree(data = tre_model_bas$fit, alpha = 0.3) +
geom_jitter(aes(color = mood), alpha = 0.7) +
scale_color_viridis_d(end = 0.8, direction = 1)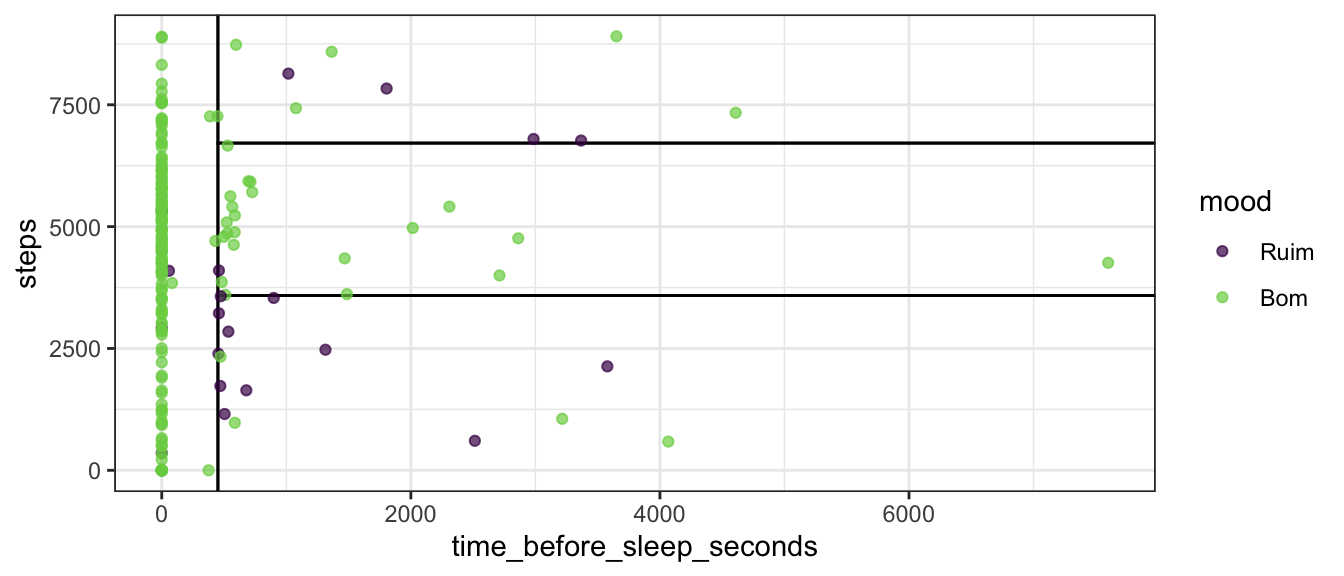
Vamos avaliar desempenho do modelo nos dados de teste:
tree_test_preds_bas %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot()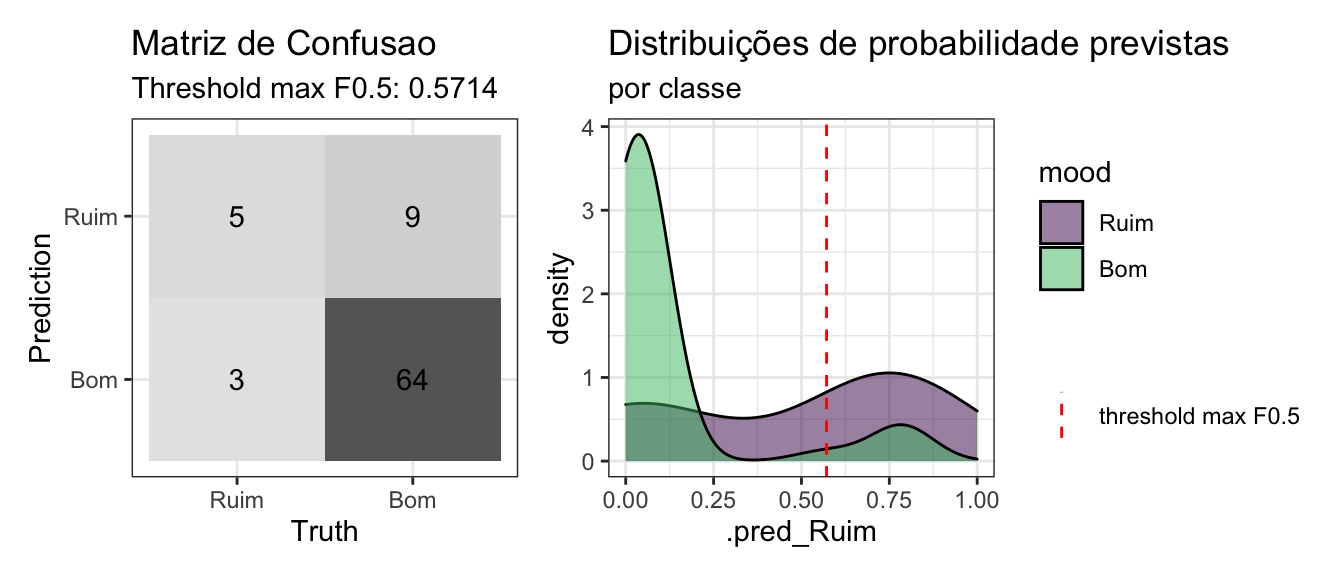
O modelo não esta muito bom… mas também não esta muito ruim para começar! 😅
Coram 5/8 acertos para classe de interesse, vamos tentar fazer o tunning deste modelo!
Tunning
Definir o modelo que será utilizado:
tree_model_tun <- decision_tree(
min_n = tune(),
cost_complexity = tune(),
tree_depth = tune()
) %>%
set_engine("rpart") %>%
set_mode("classification")
# tree_model_tun %>% translate()Definir o objeto workflow:
tree_wflow_tun <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(tree_model_tun) O grid utilizado foi alterado para tentar previnir que a árvore tenha apenas o nó raiz pois o grid default, combinado com o threshold, estava gerando um “cotoco”.
min_n: [1, 5]cost_complexity: (transformed scale): [-10, -1]tree_depth: [10, 20]
Definir um grid aleatório para otimização dos hiperparâmetros:
tree_params <-
tree_model_tun %>%
parameters() %>%
update(
min_n = min_n(c(1, 5)),
cost_complexity = cost_complexity(),
tree_depth = tree_depth(c(10, 20))
)
tree_grid <-grid_regular(tree_params, levels = 3)Ajustar modelo:
tree_res_tun <-
tree_wflow_tun %>%
tune_grid(
resamples = sleep_folds,
grid = tree_grid,
metrics = metric_set(roc_auc, mn_log_loss, pr_auc),
control = control_grid(save_pred = TRUE)
)
# Salvar cache da otimizacao
# saveRDS(tree_res_tun, "tree_res_tun.rds")Impacto de cada hiperparâmetro no resultado das métricas cálculadas para cada modelo:
(Código do gráfico)
id_best_model <-
show_best(tree_res_tun, metric = 'mn_log_loss') %>%
slice(1) %>%
pull(.config)
plot_tree_tun <-
tree_res_tun %>%
collect_metrics() %>%
mutate(best_model = if_else(.config == id_best_model,
"BestModel", "Try")
# cost_complexity = log(cost_complexity)-10
) %>%
select(.metric, mean, best_model,
cost_complexity:min_n) %>%
pivot_longer(cost_complexity:min_n,
values_to = "value",
names_to = "parameter"
) %>%
mutate(parameter = case_when(
parameter == "cost_complexity" ~ "Cost-Complexity Parameter",
parameter == "tree_depth" ~ "Tree Depth",
parameter == "min_n" ~ "Minimal Node Size",
))%>% {
ggplot(., aes(value, mean, color = best_model)) +
geom_point(alpha = 0.6, show.legend = FALSE) +
geom_point(data = subset(., best_model == 'BestModel'),
size = 4, shape = 3)+
scale_color_manual(values = c("red", "black"))+
facet_grid(.metric~parameter, scales = "free") +
labs(x = NULL, y = NULL)
}
# Codigo para mesmo grafico sem cor para melhor modelo:
# autoplot(tree_res_tun)
plot_tree_tun %>%
plotly::ggplotly()%>%
plotly::layout(showlegend = FALSE) %>%
plotly::config(displayModeBar = F)5 Melhores resultados:
show_best(tree_res_tun, metric = 'mn_log_loss') %>%
select(-.estimator, -n, -.config)## # A tibble: 5 x 6
## cost_complexity tree_depth min_n .metric mean std_err
## <dbl> <int> <int> <chr> <dbl> <dbl>
## 1 0.1 10 1 mn_log_loss 1.64 0.212
## 2 0.1 15 1 mn_log_loss 1.64 0.212
## 3 0.1 20 1 mn_log_loss 1.64 0.212
## 4 0.1 10 3 mn_log_loss 1.64 0.212
## 5 0.1 15 3 mn_log_loss 1.64 0.212Finalizar o modelo com o conjunto de parâmetros encontrados no processo de otimização:
# finalizar workflow definindo modelo final
tree_final_wflow_tun <-
finalize_workflow(
tree_wflow_tun,
select_best(tree_res_tun, metric = 'mn_log_loss') )
# Realizar ajuste final nos dados de treino
tree_final_fit_tun <- tree_final_wflow_tun %>% last_fit(sleep_split)
# Coletar previsões nos dados de teste
tree_test_preds_tun <- collect_predictions(tree_final_fit_tun)Vejamos como ficou foi o ajuste do modelo utilizando a configuração obtida no após o tunning final:
(Código do gráfico)
tre_model_tun <- pull_workflow_fit(tree_final_fit_tun$.workflow[[1]])
rattle::fancyRpartPlot(tre_model_tun$fit, sub = NULL, cex = 0.6)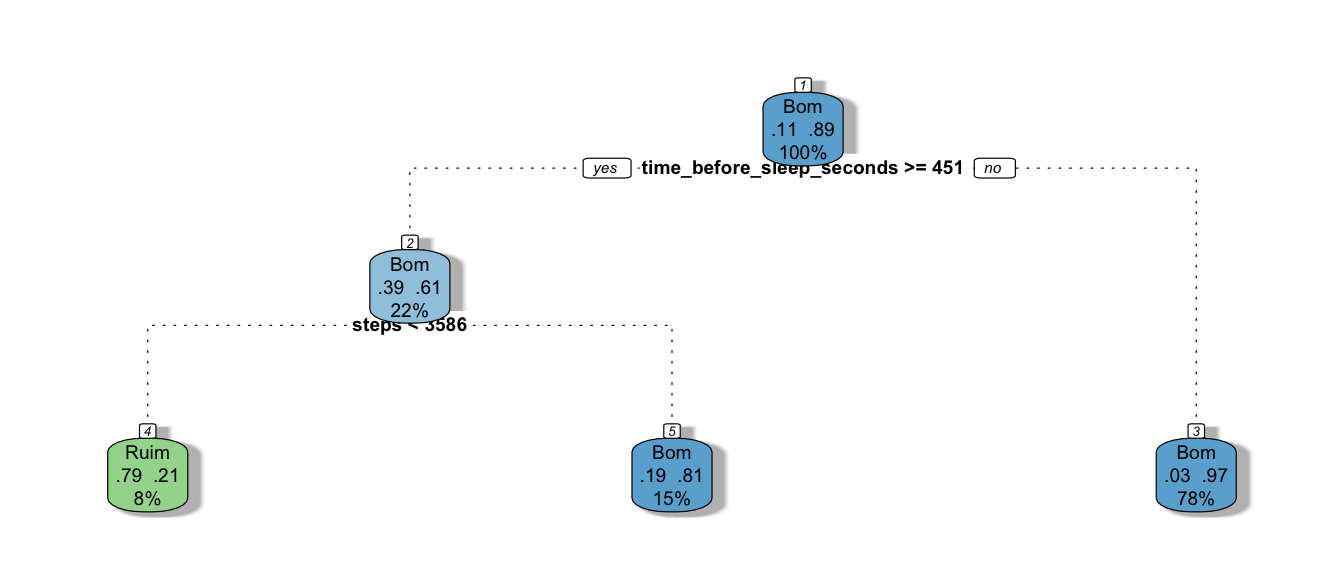
Avaliar desempenho do modelo nos dados de teste:
tree_test_preds_tun %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot()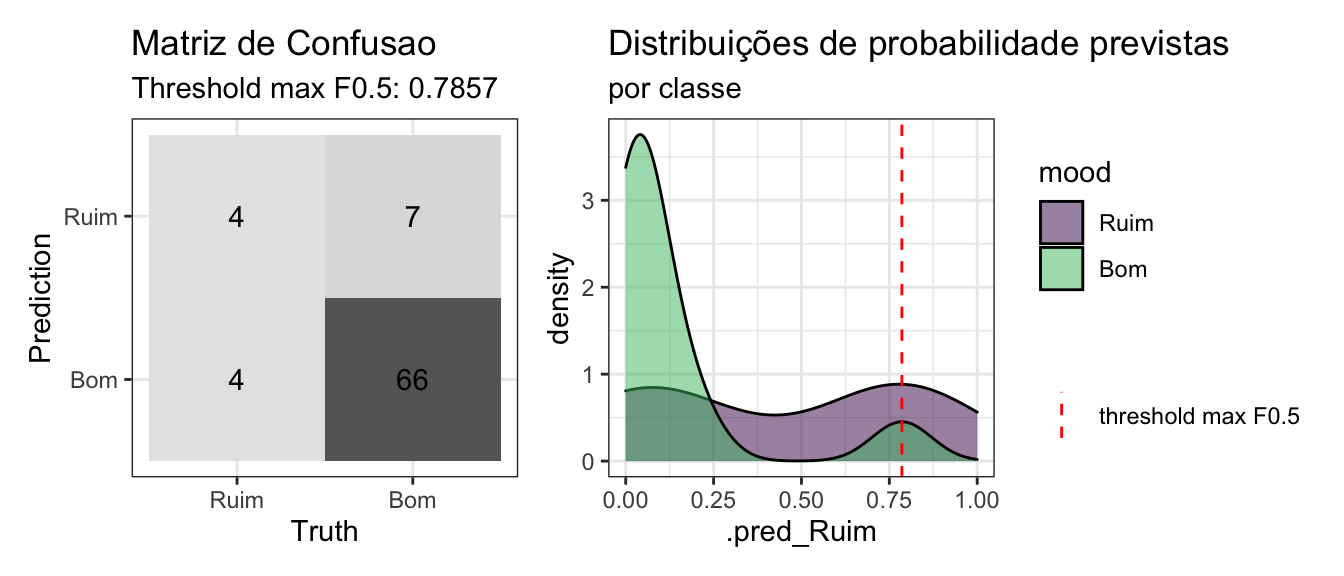
Ao comparar o modelo default com o modelo após o tunning é possível notar que o número de verdadeiros positivos foi menor porém o número de fasos positivos tbm foi menor devido ao elevado trs_fbeta encontrado (maximizando F0.5).
No geral, o modelo tunado ficou pior que o modelo default mas como o modelo de árvore de deciões costuma ser bem instável, ainda mais em um cenário de dados desbalanceados vamos apenas guardar estes resultados e dar mais um passo, combinando diversas árvore de decisões!
Random Forest
O Random Forest é um algoritmo que (de forma simplificada) realiza bootstrap em cima de árvores de decisões (modelos que utilizamos anteriormente) construindo modelos de árvores de decisões em diferentes amostras com diferentes combinações de features e assim uma previsão final é feita após uma “votação entre os modelos”.
Default
Os parâmetros default foram definidos baseados na documentação oficial do pacote ranger em https://cran.r-project.org/web/packages/ranger/ranger.pdf e o de/para para definição dos parâmetros na página do pacote parsnip em https://parsnip.tidymodels.org/reference/rand_forest.html
# raiz quadrada do numero de features
n_col = ncol(juice(sleep_recipe))
rf_model_bas <- rand_forest(
mtry = sqrt(n_col) %>% floor(), # mtry
trees = 500, # num.trees
min_n = 1 # min.node.size
) %>%
set_engine("ranger", num.threads = 4, importance = "permutation") %>%
set_mode("classification")Definir o objeto workflow:
rf_wflow_bas <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(rf_model_bas) Ajustar modelo via validação cruzada:
rf_res_bas <- fit_resamples(
rf_wflow_bas,
sleep_folds,
metrics = metric_set(roc_auc, mn_log_loss, pr_auc),
control = control_resamples(save_pred = TRUE)
)Finalizar o modelo com o conjunto de parâmetros encontrados no processo de otimização:
# Finalizar workflow com parametros selecionados (default nesse caso)
rf_final_wflow_bas <-
finalize_workflow(
rf_wflow_bas,
select_best(rf_res_bas, metric = 'mn_log_loss') )
# Realizar ajuste final nos dados de treino
rf_final_fit_bas <- rf_final_wflow_bas %>% last_fit(sleep_split)
# Coletar previsões nos dados de teste
rf_test_preds_bas <- collect_predictions(rf_final_fit_bas)Avaliar desempenho do modelo nos dados de teste:
rf_test_preds_bas %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot()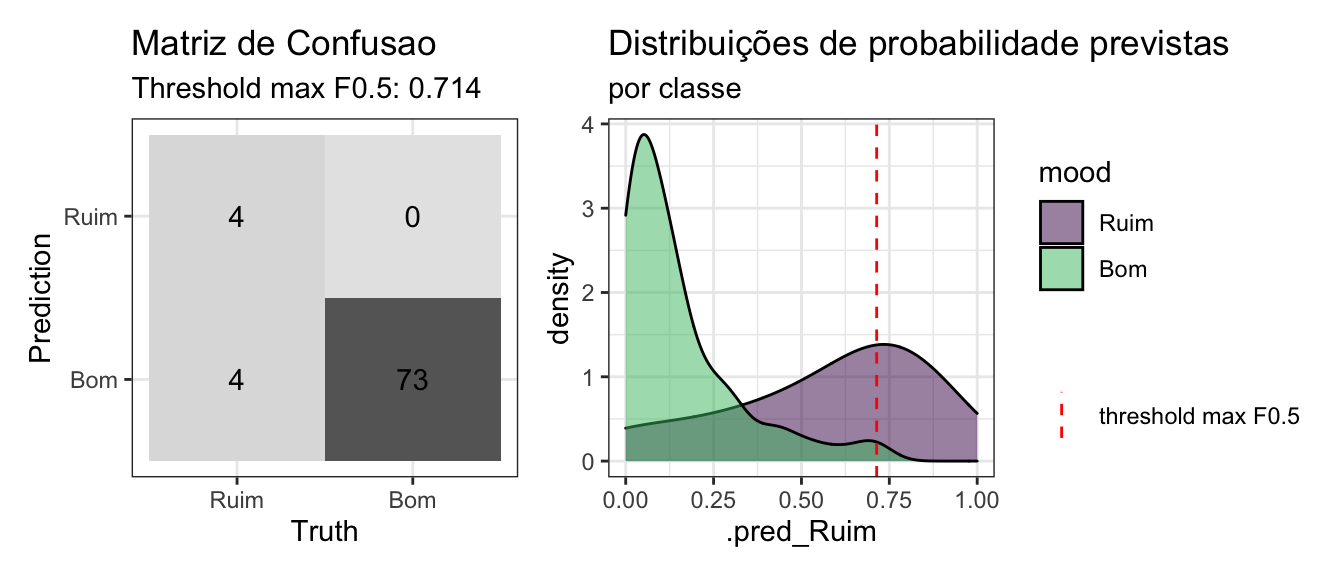
Este modelo não fez nenhuma previsão de falso positivo! Porém note que o trs_fbeta ficou bastante alto, o que deve ter ocorrido como reflexo do elevado logloss que indicaria que a incerteza que o modelo tem nas previsões esta alta.
Tunning
Definir o modelo que será utilizado:
rf_model_tun <- rand_forest(
mtry = tune(),
trees = tune(),
min_n = tune()
) %>%
set_engine("ranger", num.threads = 4, importance = "permutation") %>%
set_mode("classification")
# tree_model %>% translate()Definir o objeto workflow:
rf_wflow_tun <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(rf_model_tun) O grid utilizado tentará valores superiores e inferiores ao número de árvores default do algoritmo e vamos incluir o valor 1 ao min_n pois árvores mais longas neste método podem ser úteis. O mtry será calculado baseado nas informações do dataset de treino.
trees: [100, 900]min_n: [1, 40]mtry: [1, 20]
Definir técnica de otimização de hiperparâmetros
rf_grid <-grid_max_entropy(
trees() %>% range_set(c(100, 900)), # Default Range: [1, 2000]
min_n() %>% range_set(c(1, 40)), # Default Range: [2, 40]
finalize(mtry(), sleep_train),
size = 30)Ajustar modelo:
rf_res_tun <-
rf_wflow_tun %>%
tune_grid(
resamples = sleep_folds,
grid = rf_grid,
metrics = metric_set(roc_auc, mn_log_loss, pr_auc),
control = control_grid(save_pred = TRUE)
)
# Salvar cache da otimizacao
saveRDS(rf_res_tun, "rf_res_tun.rds")Impacto de cada hiperparâmetro no resultado das métricas cálculadas para cada modelo:
(Código do gráfico)
id_best_model <-
show_best(rf_res_tun, metric = 'mn_log_loss') %>%
slice(1) %>%
pull(.config)
plot_rf_tun <-
rf_res_tun %>%
collect_metrics() %>%
mutate(best_model = if_else(.config == id_best_model,
"BestModel", "Try")) %>%
select(.metric, mean, best_model,
mtry:min_n) %>%
pivot_longer(mtry:min_n,
values_to = "value",
names_to = "parameter"
) %>%
mutate(parameter = case_when(
parameter == "mtry" ~ "Randomly Selected Predictors",
parameter == "min_n" ~ "Minimal Node Size",
parameter == "trees" ~ "# Trees"
)) %>% {
ggplot(., aes(value, mean, color = best_model)) +
geom_point(alpha = 0.6, show.legend = FALSE) +
geom_point(data = subset(., best_model == 'BestModel'),
size = 4, shape = 3)+
scale_color_manual(values = c("red", "black"))+
facet_grid(.metric~parameter, scales = "free") +
labs(x = NULL, y = NULL)
}
# Codigo para mesmo grafico sem cor para melhor modelo:
# autoplot(rf_res_tun)
plot_rf_tun %>%
plotly::ggplotly()%>%
plotly::layout(showlegend = FALSE) %>%
plotly::config(displayModeBar = F)Melhores resultados:
show_best(rf_res_tun, metric = 'mn_log_loss') %>%
select(-.estimator, -n, -.config)## # A tibble: 5 x 6
## mtry trees min_n .metric mean std_err
## <int> <int> <int> <chr> <dbl> <dbl>
## 1 1 657 37 mn_log_loss 0.284 0.00582
## 2 1 265 32 mn_log_loss 0.284 0.00639
## 3 3 674 21 mn_log_loss 0.296 0.0190
## 4 4 448 26 mn_log_loss 0.299 0.0188
## 5 5 822 30 mn_log_loss 0.306 0.0193Finalizar o modelo com o conjunto de parâmetros encontrados no processo de otimização:
# finalizar workflow definindo modelo final
rf_final_wflow_tun <-
finalize_workflow(
rf_wflow_tun,
select_best(rf_res_tun, metric = 'mn_log_loss') )
# Realizar ajuste final nos dados de treino
rf_final_fit_tun <- rf_final_wflow_tun %>% last_fit(sleep_split)
# Coletar previsões nos dados de teste
rf_test_preds_tun <- collect_predictions(rf_final_fit_tun)Vejamos como ficou foi o ajuste do modelo utilizando a configuração obtida no após o tunning final:
Avaliar desempenho do modelo nos dados de teste:
rf_test_preds_tun %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot()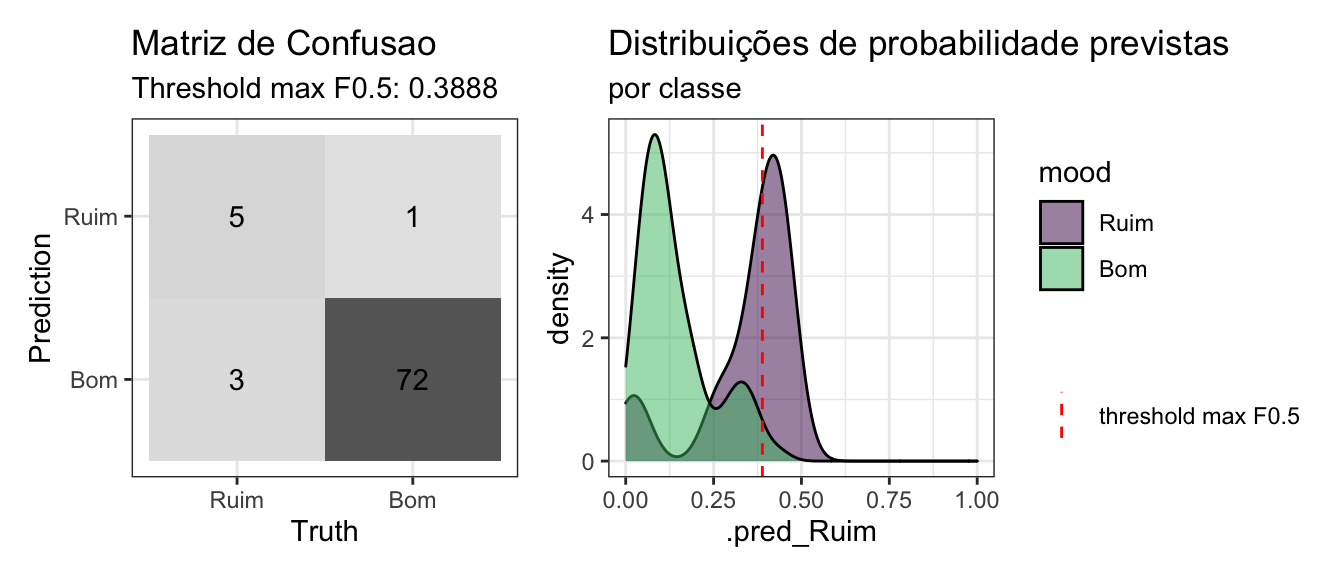
Note que apesar do maior número de Verdadeiros Positivos, este modelo apresentou um Falso Positivo. Parece estranho pois é exatamente o que queriamos evitar porém é possível notar que o logloss foi bem inferior e o trs_fbeta está bem mais razoavel agora.
Importancia de cada feature conforme o modelo:
vip::vip(pull_workflow_fit(rf_final_fit_tun$.workflow[[1]]))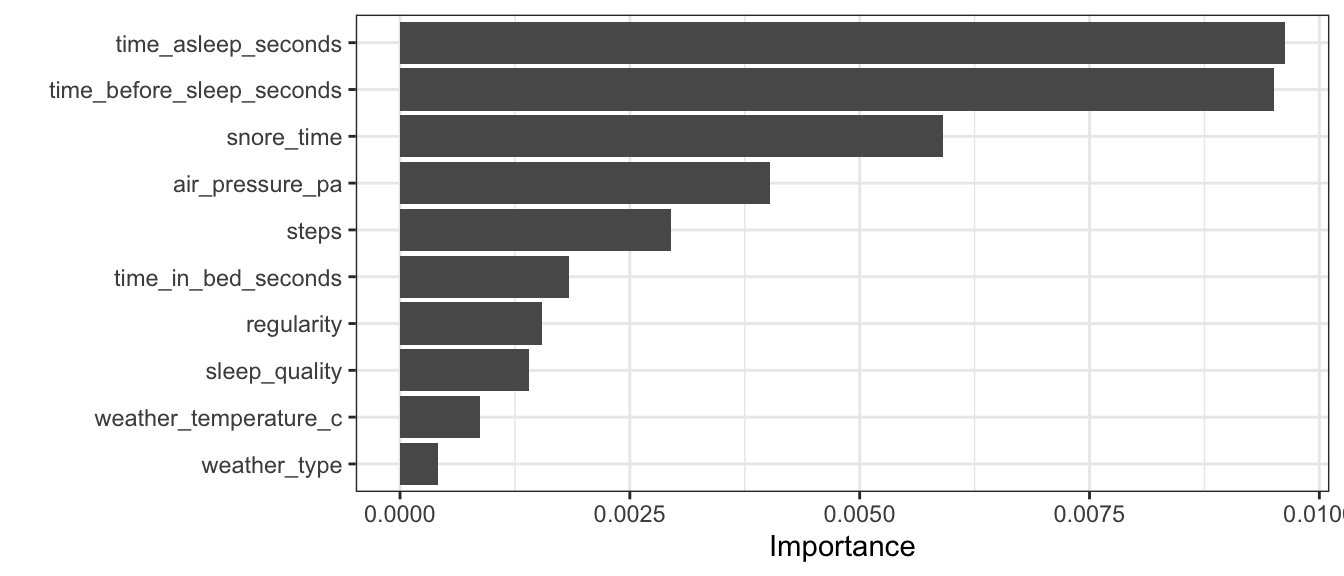
Diferente do modelo baseado em 1 unica árvore de decisões, a feature steps não foi tão importante assim. A time_asleep_seconds foi a mais importante mas com a ordem de grandeza muito próxima de time_before_sleep_seconds.
Random Forest é um excelente modelo e poderíamos investir mais tempo tentando otimizando sua performance mas para este post acho que já esta suficiente. Vamos para o próximo modelo! 😍
LightGBM
Este modelo consiste em um método de boosting. Também é baseado nos modelos de árvore de decisões, mas, diferentemente do Random Forest, suas árvores são calculadas em sequência, “aprendendo” com o erro das árvores anteriores.
A mecânica do LightGBM é um pouco diferente do XGBoost. Não entrarei em detalhes sobre a teoria neste post até porque a documentação oficial no github em https://github.com/microsoft/LightGBM é bastante rica, e seus recursos são muito bem apresentados neste link: https://github.com/microsoft/LightGBM/blob/master/docs/Features.rst
Links úteis para consulta ao trabalhar com este algoritmo:
- Documentação oficial: https://lightgbm.readthedocs.io/en/latest/
- Excelente post: https://machinelearningmastery.com/light-gradient-boosted-machine-lightgbm-ensemble/
- Documentação oficial do pacote
treesnip: https://curso-r.github.io/treesnip/articles/working-with-lightgbm-catboost.html - Repositório no github do pacote
treesnip: https://github.com/curso-r/treesnip - Ótimo link para consulta dos parâmetros: https://sites.google.com/view/lauraepp/parameters
Default
Os parâmetros default foram definidos baseados na documentação oficial do pacote lightgbm em https://lightgbm.readthedocs.io/en/latest/ e o de/para para definição dos parâmetros na página do (incrível 🤩) pacote treesnip em https://github.com/curso-r/treesnip/blob/master/R/lightgbm.R
lgbm_model_bas <- parsnip::boost_tree(
mode = "classification",
trees = 100, # num_iterations
learn_rate = 0.1, # fixo
min_n = 20, # min_data_in_leaf
tree_depth = 6, # max_depth
sample_size = 1, # bagging_fraction
mtry = 1, # feature_fraction
loss_reduction = 0 # min_gain_to_split
) %>%
set_engine("lightgbm",
nthread = 4,
importance = "permutation") %>%
set_mode("classification")Definir o objeto workflow:
lgbm_wflow_bas <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(lgbm_model_bas) Ajustar modelo via validação cruzada:
lgbm_res_bas <- fit_resamples(
lgbm_wflow_bas,
sleep_folds,
metrics = metric_set(roc_auc, mn_log_loss, pr_auc),
control = control_resamples(save_pred = TRUE)
)
saveRDS(lgbm_res_bas, "lgbm_res_bas.rds")Finalizar o modelo com o conjunto de parâmetros encontrados no processo de otimização:
# Finalizar workflow com parametros selecionados (default nesse caso)
lgbm_final_wflow_bas <-
finalize_workflow(
lgbm_wflow_bas,
select_best(lgbm_res_bas, metric = 'mn_log_loss') )
# Realizar ajuste final nos dados de treino
lgbm_final_fit_bas <- lgbm_final_wflow_bas %>% last_fit(sleep_split)
# Coletar previsões nos dados de teste
lgbm_test_preds_bas <- collect_predictions(lgbm_final_fit_bas)Avaliar desempenho do modelo nos dados de teste:
lgbm_test_preds_bas %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot()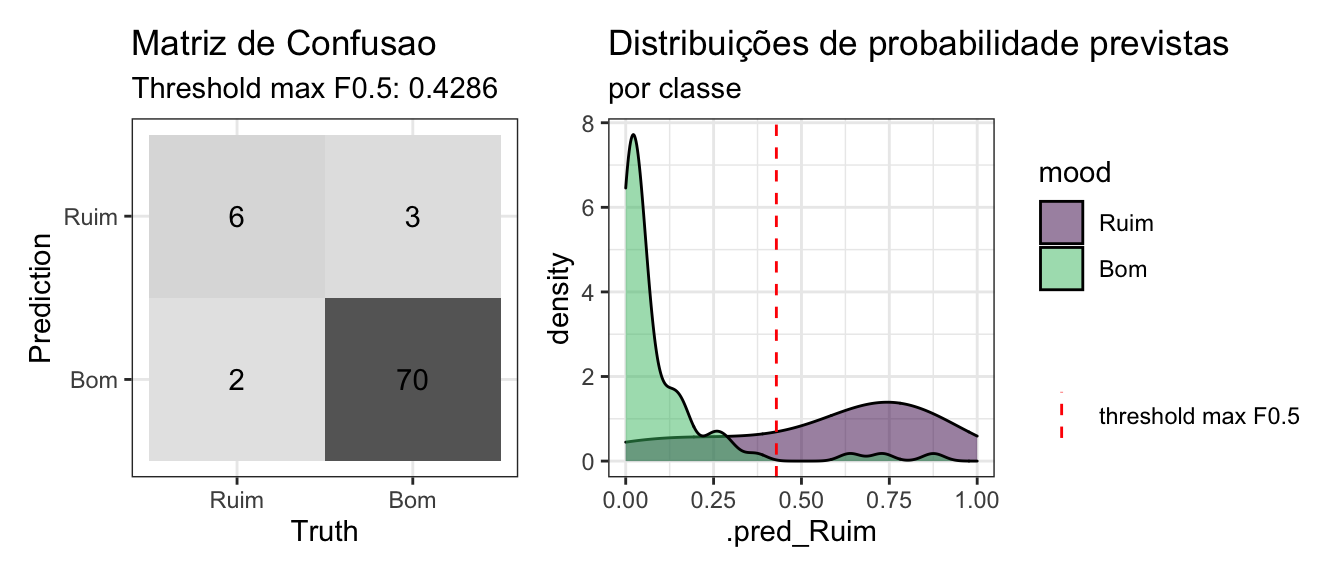
Tunning
Para o tunning vamos utilizar uma estratégia um pouco diferente. Vamos fixar o número de árvores trees e a taxa de aprendizado learning_rate pois vamos separar mais uma pequena parte dos dados para usar o recurso early_stopping. Esta opção basicamente “trava” o crescimento da árvore caso o modelo não melhore a performance a partir da n-ésima iteração.
lgbm_model_tun <- parsnip::boost_tree(
mode = "classification",
trees = 700, # autotune com early stopping
learn_rate = 0.01, # early stopping
min_n = tune(), # min_data_in_leaf
tree_depth = tune(), # max_depth
sample_size = 1, # bagging_fraction, n funciona com goss
mtry = tune(), # feature_fraction
loss_reduction = tune() # min_gain_to_split
) %>%
set_engine("lightgbm", nthread = 4,
# parametros para early stopping
early_stop = 30,
validation = .20,
eval_metric = "mn_log_loss",
importance = "permutation"
# feature_fraction = tune("feature_fraction")
) %>%
set_mode("classification")
# tree_model %>% translate()Definir o objeto workflow:
lgbm_wflow_tun <- workflow() %>%
add_recipe(sleep_recipe) %>%
add_model(lgbm_model_tun) Definir grid para otimização de hiperparâmetros baseados nas sugestões de github/Laurae2 em uma issue no repositório oficial do modelo
lightgbm_params <-
dials::parameters(
# learn_rate(), # learning_rate
# trees() # num_iterations
min_n(), # min_data_in_leaf
tree_depth(c(2, 63)), # max_depth
# sample_prop(c(0.4, 1)), # bagging_fraction (vai para sample_size)
mtry(), # feature_fraction
loss_reduction() # min_gain_to_split
)
lgbm_grid <- lightgbm_params %>%
finalize(sleep_train) %>%
grid_max_entropy(size = 30)Ajustar modelo:
lgbm_res_tun <-
lgbm_wflow_tun %>%
tune_grid(
resamples = sleep_folds,
grid = lgbm_grid,
metrics = metric_set(roc_auc, mn_log_loss, pr_auc),
control = control_grid(save_pred = TRUE)
)
# Salvar cache da otimizacao
saveRDS(lgbm_res_tun, "lgbm_res_tun.rds")Impacto de cada hiperparâmetro no resultado das métricas cálculadas para cada modelo:
(Código do gráfico)
id_best_model <-
show_best(lgbm_res_tun, metric = 'mn_log_loss')[1, ] %>%
pull(.config)
plot_lgbm_tun <-
lgbm_res_tun %>%
collect_metrics() %>%
mutate(best_model = if_else(.config == id_best_model,
"BestModel", "Try")) %>%
select(.metric, mean, best_model,
mtry:loss_reduction) %>%
pivot_longer(mtry:loss_reduction,
values_to = "value",
names_to = "parameter"
) %>% {
ggplot(., aes(value, mean, color = best_model)) +
geom_point(alpha = 0.6, show.legend = FALSE) +
geom_point(data = subset(., best_model == 'BestModel'),
size = 4, shape = 3)+
scale_color_manual(values = c("red", "black"))+
facet_grid(.metric~parameter, scales = "free") +
labs(x = NULL, y = NULL)
}
# Codigo para mesmo grafico sem cor para melhor modelo:
# autoplot(lgbm_res_tun)
plot_lgbm_tun %>%
plotly::ggplotly()%>%
plotly::layout(showlegend = FALSE) %>%
plotly::config(displayModeBar = F)Melhores resultados:
show_best(lgbm_res_tun, metric = 'mn_log_loss') %>%
select(-.estimator, -n, -.config)## # A tibble: 5 x 7
## mtry min_n tree_depth loss_reduction .metric mean std_err
## <int> <int> <int> <dbl> <chr> <dbl> <dbl>
## 1 2 35 28 0.000000000108 mn_log_loss 0.259 0.00888
## 2 1 16 5 0.00000160 mn_log_loss 0.267 0.0100
## 3 4 28 31 0.000507 mn_log_loss 0.272 0.0111
## 4 5 39 62 0.000164 mn_log_loss 0.273 0.00955
## 5 2 14 19 0.0642 mn_log_loss 0.274 0.0138Finalizar o modelo com o conjunto de parâmetros encontrados no processo de otimização:
# finalizar workflow definindo modelo final
lgbm_final_wflow_tun <-
finalize_workflow(
lgbm_wflow_tun,
select_best(lgbm_res_tun, metric = 'mn_log_loss') )
# Realizar ajuste final nos dados de treino
lgbm_final_fit_tun <- lgbm_final_wflow_tun %>% last_fit(sleep_split)
# Coletar previsões nos dados de teste
lgbm_test_preds_tun <- collect_predictions(lgbm_final_fit_tun)Vejamos como ficou foi o ajuste do modelo utilizando a configuração obtida no após o tunning final:
Avaliar desempenho do modelo nos dados de teste:
lgbm_test_preds_tun %>%
mutate(mood = factor(mood, levels = c("Ruim", "Bom"), ordered = TRUE)) %>%
conf_mat_plot()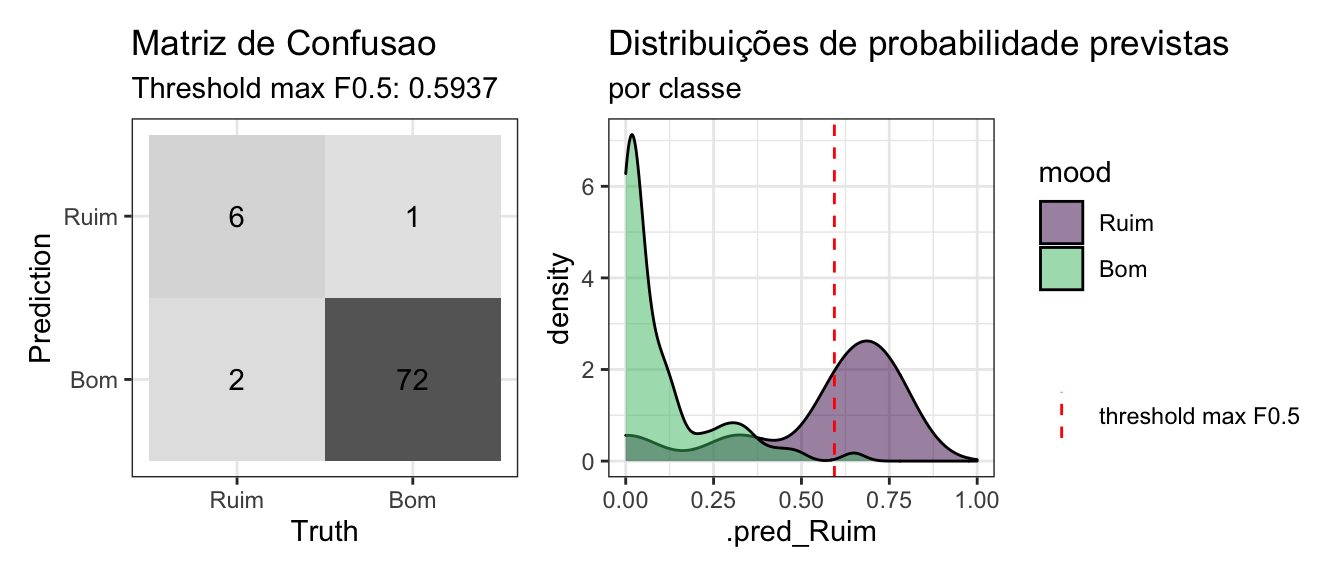
Que maravilha! Modelos acertaram mais a classe de interesse do que os anteriores (apesar do default ainda apresentar alta proporção de falsos positivos). Note ainda que o LightGBM após o tunning apresentou as melhores métricas no geral (melhor AUC-PR, menor logloss e um bom equilíbrio no trade-off de Precision x Recall).
Vejamos quais as features mais importantes no ajuste do modelo:
lgbm_imp_tun <- lightgbm::lgb.importance(lgbm_final_fit_tun$.workflow[[1]]$fit$fit$fit, percentage = T)
lgbm_imp_tun%>%
mutate(Feature = reorder(Feature, Gain)) %>%
ggplot(aes(x = Feature, y = Gain))+
geom_bar(stat = "identity")+
labs(y = "Importance", x= "")+
coord_flip()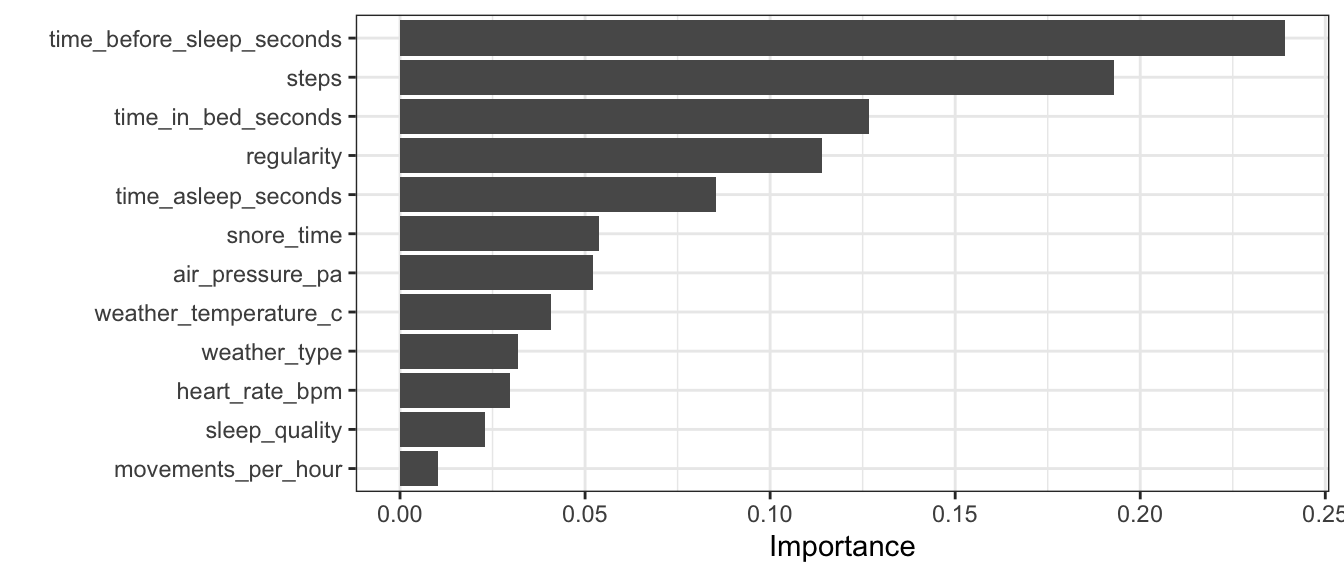
Seleção do modelo 🤔
Comparar os modelos de forma visual com os gráficos da ROC AUC e da PR AUC:
(Código do gráfico)
auc_plots <-
bind_rows(
null_test_preds_bas %>% mutate(model = "null baseline"),
tree_test_preds_bas %>% mutate(model = "rpart default"),
tree_test_preds_tun %>% mutate(model = "rpart tunning"),
rf_test_preds_bas %>% mutate(model = "rf default"),
rf_test_preds_tun %>% mutate(model = "rf tunning"),
lgbm_test_preds_bas %>% mutate(model = "lgbm default"),
lgbm_test_preds_tun %>% mutate(model = "lgbm tunning")
) %>%
plot_auc() +
plot_annotation(title = 'Resultados nos dados de teste',
theme = theme(plot.title = element_text(hjust = 0.4)))
auc_plots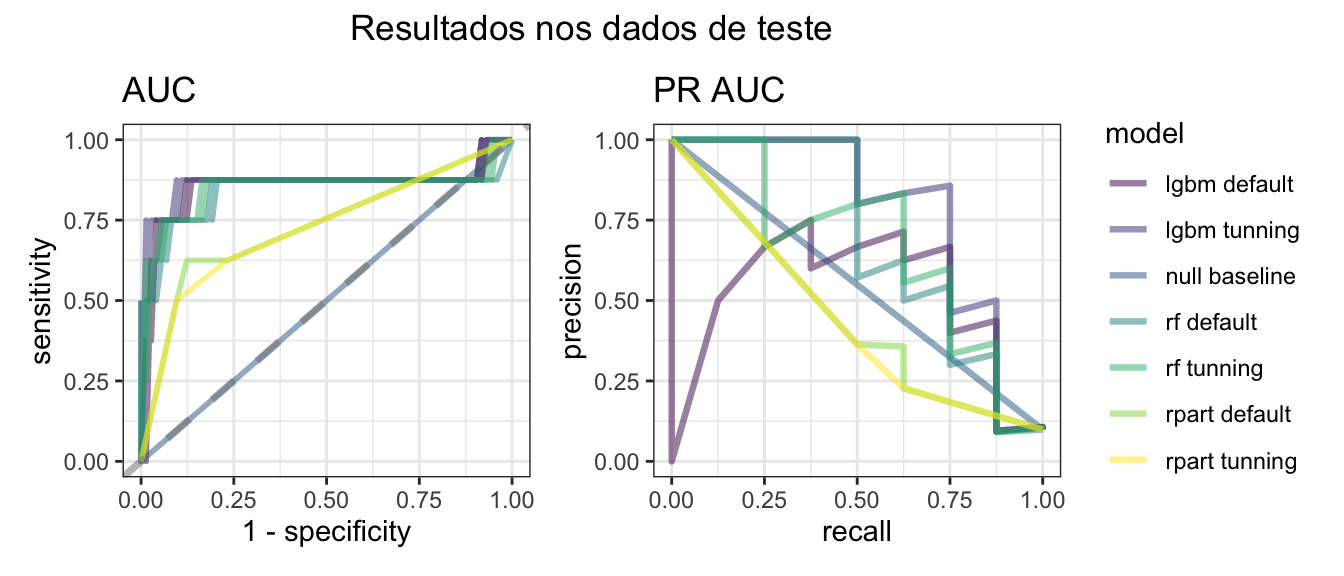
Apenas olhando o gráfico não da para fazer uma análise conclusiva, vejamos as medidas de qualidade (ordenado por auc_pr):
(Código da tabela)
test_results <-
bind_rows(
evalue_model(null_test_preds_bas, model = "null baseline", null_model = TRUE),
evalue_model(tree_test_preds_bas, model = "rpart default"),
evalue_model(tree_test_preds_tun, model = "rpart tunning"),
evalue_model(rf_test_preds_bas, model = "rf default"),
evalue_model(rf_test_preds_tun, model = "rf tunning"),
evalue_model(lgbm_test_preds_bas, model = "lgbm default"),
evalue_model(lgbm_test_preds_tun, model = "lgbm tunning")
) %>% print_table(round = 4, evalue_model = T)
test_resultsO modelo LightGBM após o processo de tunning foi o que apresentou as melhores medidas no geral. Note que o LightGBM com os parâmetro default ficou pior do que o modelo nulo 😱! Isso mostra como o processo de tunning pode ser importante. Além disso note que o modelo rf baseline apresentou o segundo maior AUC-PR mas o pior logloss (note que o threshold está muito alto e as demais métricas não ficaram muito boas).
Portanto, apenas os modelos LightGBM e Random Forest apresentaram resultados melhores que um modelo nulo (sempre estima a classe majoritária) e como o LightGBM foi o mais satisfatório, este será o modelo selecionado. 😎
Previsão em dados novos 💫
Obter as previsões nos novos dados:
trs_final <- evalue_model(lgbm_test_preds_tun, model = "lgbm tunning")$trs_fbeta
final <-
predict(lgbm_final_fit_tun$.workflow[[1]], new_sleep, type = "prob") %>%
mutate(.pred_class = ifelse(.pred_Ruim >= trs_final, "Ruim", "Bom"))
# new_sleep %>% filter(final$.pred_class == "Ruim")Comparar a quantidade de previsões de cada classe com o conjunto de treino/teste:
(Código para tabela abaixo)
tab <-
full_join(sleep_train %>% count(mood) %>% mutate(prop = n/sum(n)*100),
sleep_test %>% count(mood) %>% mutate(prop = n/sum(n)*100),
by = "mood") %>%
full_join(final %>%
count(mood = .pred_class) %>% mutate(prop = n/sum(n)*100)) %>%
print_table(round = 2,
columns = list(
n.x = colDef(name = "N"),
prop.x = colDef(name = "(%)", align = "left"),
n.y = colDef(name = "N"),
prop.y = colDef(name = "(%)", align = "left"),
n = colDef(name = "N"),
prop = colDef(name = "(%)", align = "left")
),
columnGroups = list(
colGroup(name = "Train", columns = c("n.x", "prop.x")),
colGroup(name = "Test", columns = c("n.y", "prop.y")),
colGroup(name = "New Data", columns = c("n", "prop"))
))
tab(Código do cálculo das medidas abaixo)
# ref: https://en.wikipedia.org/wiki/Sensitivity_and_specificity
# Obter medidas da matriz de confusao
tp = evalue_model(lgbm_test_preds_tun, model = "lgbm tunning")$tp
tn = evalue_model(lgbm_test_preds_tun, model = "lgbm tunning")$tn
fn = evalue_model(lgbm_test_preds_tun, model = "lgbm tunning")$fn
fp = evalue_model(lgbm_test_preds_tun, model = "lgbm tunning")$fn
# true positive rate
tpr = tp / (tp + fn)
# false negative rate
fnr = 1 - tpr
#false positive rate
fpr = fp / (fp + tn)
Como nosso modelo foi otimizado para ser menos “alarmista” (com uma Taxa de Falso Positivo: 2.7%) é possível que o modelo tenha deixado passar alguns dias em que mood=="Ruim" (Taxa de Falso Negativo: 25%). Não vejo isto como um grande problema pois dado a pequena quantidade de dados disponíveis até que o resultado para a classe de interesse estava bem razoável (Taxa de Verdadeiro Positivo: 75%).
Para não alongar aida mais o post com análise exploratória das previsões, vamos comparar como foram as previsões nestes novos dados em relação aos dados utilizados para treinar o modelo e ver se, pelo menos visualmente, o modelo esteja conseguindo prever de semelhante ao padrão de dados conhecidos.
A técnica UMAP será utilizada com a finalidade de reduzir a dimensionalidade para visualização:
(Código para gráfico abaixo)
# Treinar UMAP:
sleep_umap <- juice(sleep_recipe) %>% select(-mood) %>% umap::umap()
# Aplicar em novos dados:
new_data <- bake(sleep_recipe, new_sleep) %>% select(-mood)
new_data_umap <- predict(sleep_umap, new_data)
# Preparar plot comparando treino com novos dados:
umap_plot <-
bind_rows(
sleep_umap$layout %>%
as_tibble() %>%
bind_cols(juice(sleep_recipe) %>% select(mood)) %>%
bind_cols(dataset = "Train")
,
new_data_umap %>%
as_tibble() %>%
mutate(mood = factor(final$.pred_class,
levels = c("Ruim", "Bom"))) %>%
bind_cols(dataset = "New Data")
) %>%
mutate(dataset = factor(dataset, levels = c("New Data", "Train"))) %>% {
ggplot(., aes(x = V1, y = V2, color = mood, shape = mood))+
geom_point(show.legend = F)+
geom_point(aes(x = V1, y = V2, color = mood),
data = subset(., mood == 'Ruim'),
size = 2, shape = 3)+
labs(x = "", y = "",
title = "UMAP (Uniform Manifold Approximation and Projection)")+
scale_color_viridis_d(end = 0.8, direction = 1)+
# scale_size_manual(values=c(2,5))+
theme(legend.position = "bottom")+
facet_wrap(~dataset)
}
umap_plot %>%
plotly::ggplotly()%>%
plotly::layout(showlegend = FALSE) %>%
plotly::config(displayModeBar = F)Parece que o modelo fez previsões nos novos dados em um padrão específico dos dados (à direita) enquanto que nos dados de treino podemos observar alguma informação da classe Ruim na massa de dados à esquerda. Isso pode estar acontecendo devido ao foco que demos para minimizar falsos positivos. É uma boa indicação para analisar melhor o padrão que o modelo esta aprendendo em relação aos falsos negativos.
Conclusão 🍻
Apesar da pequena quantidade dados dados disponíveis, conseguimos ajustar um modelo razoável para prever a qualidade de sono em dias que não foram registrados!
Utilizamos diversas técnicas de Machine Leaning combinadas em dados reais (que não são nada comportados) e, obviamente, para colocar um modelo em produção na vida real seria necessário aplicar mais uma série de análises, além de entender como o modelo está funcionando, aplicando técnicas de XAI (Explainable AI) mas isso pode ser assunto para um futuro post, hora de dormir! 😴
Espero que este pequeno “case” seja útil para você! Para mim foi ótimo combinar a prática do uso do pacote tidymodels para resolver um problema com dados reais com um estudo que me trouxe mais auto-conhecmento e um monte de insights pessoais.
Referências 🧳
- https://juliasilge.com/blog/wind-turbine/
- https://juliasilge.com/blog/hotels-recipes/
- https://juliasilge.com/blog/xgboost-tune-volleyball/
- http://www.rebeccabarter.com/blog/2020-03-25_machine_learning/
- https://machinelearningmastery.com/imbalanced-classification-with-python/
- https://machinelearningmastery.com/threshold-moving-for-imbalanced-classification/
- https://machinelearningmastery.com/undersampling-algorithms-for-imbalanced-classification/
- https://machinelearningmastery.com/fbeta-measure-for-machine-learning/
- https://sites.google.com/view/lauraepp/parameters
- https://github.com/microsoft/LightGBM/issues/695
https://www.usp.br/espacoaberto/?materia=a-importancia-de-dormir-bem↩︎
https://machinelearningmastery.com/tour-of-evaluation-metrics-for-imbalanced-classification/↩︎
https://machinelearningmastery.com/fbeta-measure-for-machine-learning/↩︎
https://machinelearningmastery.com/undersampling-algorithms-for-imbalanced-classification/↩︎
https://machinelearningmastery.com/roc-curves-and-precision-recall-curves-for-imbalanced-classification/↩︎































Share this post
Twitter
LinkedIn
Email